Zmiana indeksów w sekwencjonowaniu DNA: cicha groźba dla integralności danych i jak nowoczesne technologie z nią walczą. Odkryj wpływ, wykrywanie i przyszłość tego krytycznego zjawiska. (2025)
- Wprowadzenie: Czym jest zmiana indeksów w sekwencjonowaniu DNA?
- Kontekst historyczny i odkrycie zmiany indeksów
- Mechanizmy stojące za zmianą indeksów: Jak i dlaczego to występuje
- Czynniki technologiczne: platformy sekwencjonowania i protokoły
- Konieczności dla dokładności danych genomicznych i badań
- Metody wykrywania: identyfikacja i kwantyfikacja zmiany indeksów
- Strategie łagodzenia: rozwiązania laboratoryjne i bioinformatyczne
- Standardy branżowe i wytyczne (np. Illumina, NIH)
- Rynek i zainteresowanie opinii publicznej: aktualne trendy i prognozy na 5 lat
- Przyszłe spojrzenie: innowacje, wyzwania i droga naprzód
- Źródła i odniesienia
Wprowadzenie: Czym jest zmiana indeksów w sekwencjonowaniu DNA?
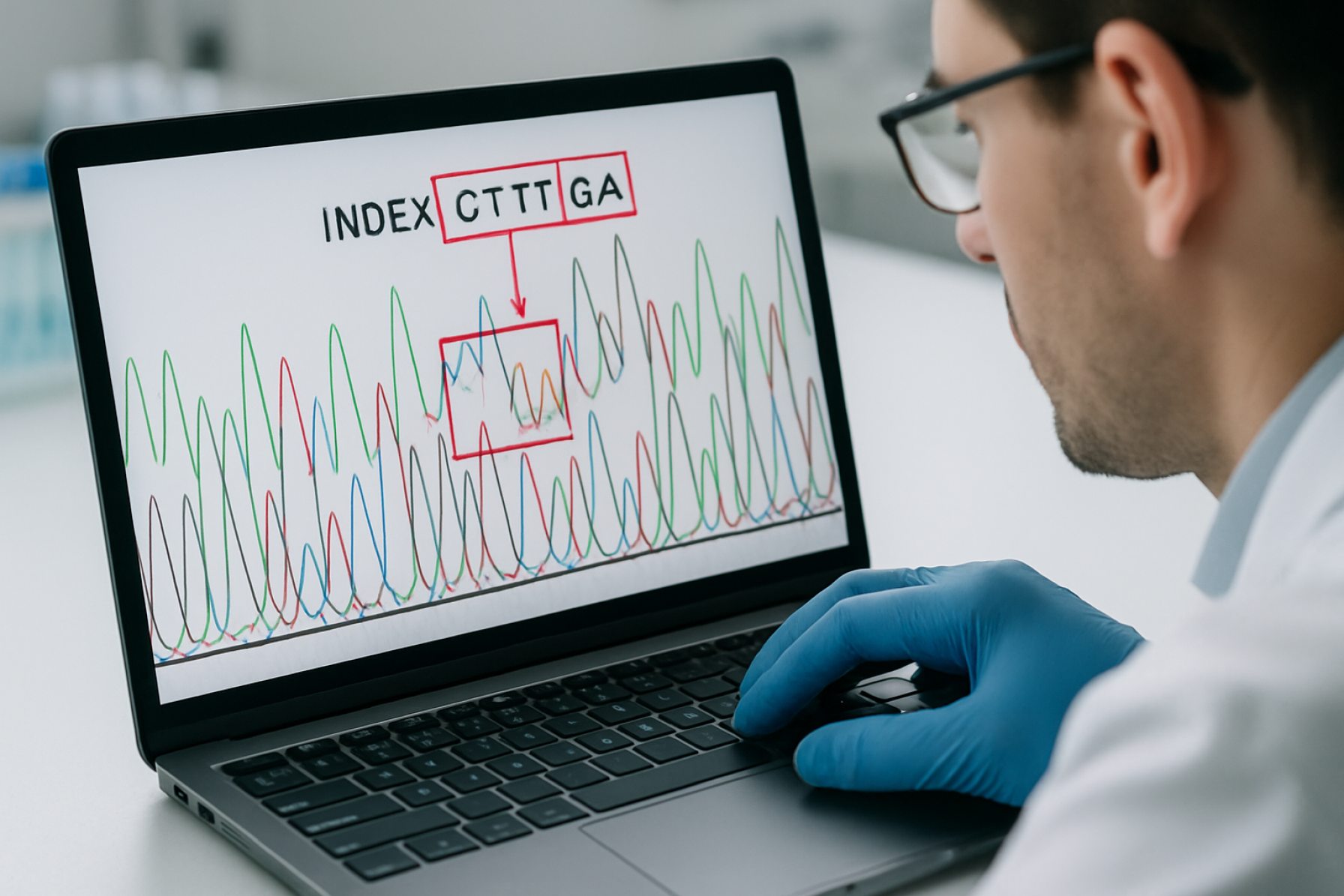
Zmiana indeksów, znana również jako „skakanie indeksów”, to zjawisko obserwowane w platformach sekwencjonowania DNA o wysokiej przepustowości, szczególnie tych, które wykorzystują strategie sekwencjonowania multiplikowanego. W tych podejściach unikalne krótkie sekwencje DNA – nazywane indeksami lub kodami kreskowymi – są dołączane do pojedynczych próbek DNA. To pozwala na połączenie wielu próbek i sekwencjonowanie ich razem w jednym cyklu, z oczekiwaniem, że każde odczytanie będzie później dokładnie przypisane do swojej oryginalnej próbki na podstawie indeksu. Jednak zmiana indeksów występuje, gdy sekwencja indeksu związana z fragmentem DNA jest nieprawidłowo przypisywana, co skutkuje błędnym przypisaniem odczytów do niewłaściwej próbki.
Ten błąd może wystąpić w różnych etapach procesu sekwencjonowania, w tym podczas przygotowania bibliotek, generacji klastrów i samego sekwencjonowania. Problem ten jest szczególnie wyraźny w platformach, które używają komórek przepływowych z wzorami i amplifikacji z wykluczeniem, takich jak te opracowane przez Illumina, Inc., wiodącego dostawcę technologii sekwencjonowania nowej generacji (NGS). W tych systemach swobodnie unoszące się adaptery lub niekompletne zdarzenia ligacji mogą prowadzić do transferu sekwencji indeksów między fragmentami DNA, powodując, że część odczytów będzie miała błędny indeks.
Konsekwencje zmiany indeksów są znaczące dla wielu zastosowań sekwencjonowania DNA. W badaniach, gdzie istotne są rzadkie warianty lub sygnały o niskiej obfitości – takich jak genomika pojedynczych komórek, metagenomika lub diagnostyka kliniczna – nawet niewielka stawka błędnego przypisania indeksów może prowadzić do fałszywych pozytywów, zanieczyszczeń lub błędnych wniosków. W miarę jak przepustowość sekwencjonowania i poziomy multiplikacji rosną, potencjalny wpływ zmiany indeksów na jakość i interpretację danych staje się coraz bardziej widoczny.
Świadomość dotycząca zmiany indeksów skłoniła społeczność genomową do opracowania zarówno strategii eksperymentalnych, jak i obliczeniowych w celu złagodzenia jej skutków. Obejmuje to użycie unikalnych indeksów podwójnych (UDIs), ulepszonych protokołów przygotowania bibliotek oraz metod bioinformatycznego filtrowania. Organizacje takie jak Narodowe Instytuty Zdrowia (NIH) oraz Narodowy Instytut Badań Genomu Ludzkiego (NHGRI) podkreśliły znaczenie dokładnej identyfikacji próbek w badaniach sekwencjonowania, zwracając uwagę na potrzebę solidnych rozwiązań w celu zmniejszenia zmiany indeksów.
Podsumowując, zmiana indeksów jest istotnym wyzwaniem technicznym we współczesnym sekwencjonowaniu DNA, ze skutkami dla integralności danych, powtarzalności i wiarygodności wyników naukowych oraz klinicznych. Zrozumienie jej mechanizmów oraz opracowywanie skutecznych przeciwdziałań pozostaje aktywnym obszarem badań i innowacji w dziedzinie genomiki.
Kontekst historyczny i odkrycie zmiany indeksów
Zjawisko zmiany indeksów, znane również jako „skakanie indeksów”, pojawiło się jako istotny problem w dziedzinie sekwencjonowania DNA o wysokiej przepustowości w połowie lat 2010-tych. Zmiana indeksów odnosi się do błędnego przypisywania indeksów próbek (kodów kreskowych) podczas multiplikowanych cykli sekwencjonowania, co skutkuje, że odczyty są nieprawidłowo przypisywane do niewłaściwej próbki. Ten artefakt może kompromitować dokładność analiz opartych na sekwencjach, szczególnie w zastosowaniach wymagających wysokiej czułości, takich jak genomika pojedynczych komórek i metagenomika.
Kontekst historyczny zmiany indeksów jest ściśle powiązany z szybkim rozwojem technologii sekwencjonowania nowej generacji (NGS). Wczesne platformy NGS, takie jak te opracowane przez Illumina i Thermo Fisher Scientific, umożliwiły jednoczesne sekwencjonowanie wielu próbek poprzez przyłączenie unikalnych sekwencji indeksów do każdej biblioteki. To podejście multiplikujące znacznie zwiększyło przepustowość i obniżyło koszty, ale wprowadziło również nowe źródła błędów. Początkowo skupiano się na minimalizacji kontaminacji między próbkami podczas przygotowania bibliotek i sekwencjonowania. Jednak w miarę poprawy głębokości i czułości sekwencjonowania, badacze zaczęli dostrzegać niespodziewane wzorce błędnych przypisań odczytów, które nie mogły być wyjaśnione przez tradycyjną kontaminację.
Odkrycie zmiany indeksów jako wyraźnego artefaktu technicznego po raz pierwszy zostało systematycznie opisane w 2017 roku, gdy badania wykorzystujące wzorowane platformy komórkowe Illumina (takie jak HiSeq 4000 i NovaSeq) zgłaszały podwyższone wskaźniki błędnego przypisania indeksów. Naukowcy odkryli, że zastosowanie chemii amplifikacji z wykluczeniem (ExAmp), która zastąpiła amplifikację mostkową w tych nowszych platformach, było związane ze zwiększoną liczbą zdarzeń zmiany indeksów. Przypisano to obecności swobodnie unoszących się adapterów oraz fizycznej bliskości klastrów na wzorowanych komórkach przepływowych, co ułatwiało transfer sekwencji indeksów między bibliotekami podczas generacji klastrów. Problem ten był szczególnie widoczny w eksperymentach RNA-seq pojedynczych komórek, gdzie nawet niskie poziomy zmiany indeksów mogły prowadzić do znaczących artefaktów danych.
W odpowiedzi na te ustalenia dostawcy technologii sekwencjonowania, tacy jak Illumina, przyznali się do problemu i zaczęli zalecać najlepsze praktyki w celu łagodzenia zmiany indeksów, w tym stosowanie unikalnych indeksów podwójnych i ulepszonych protokołów czyszczenia bibliotek. Szeroka społeczność genomowa, w tym organizacje takie jak Narodowy Instytut Badań Genomu Ludzkiego (NHGRI), podkreśliła znaczenie zrozumienia i kontrolowania zmiany indeksów w projektowaniu eksperymentalnym i interpretacji danych. W 2025 r. prowadzone są dalsze badania, które mają na celu dopracowanie chemii sekwencjonowania i podejść informatycznych, aby jeszcze bardziej zredukować wpływ zmiany indeksów na badania genomiczne.
Mechanizmy stojące za zmianą indeksów: Jak i dlaczego to występuje
Zmiana indeksów, znana również jako skakanie indeksów, to zjawisko w sekwencjonowaniu DNA o wysokiej przepustowości, w którym specyficzne dla próbki sekwencje indeksów (kody kreskowe) przypisane podczas przygotowania bibliotek są błędnie związane z fragmentami DNA z innych próbek. To błędne przypisanie może prowadzić do kontaminacji między próbkami odczytów sekwencjonowania, potencjalnie wpływając na dalsze analizy, szczególnie w eksperymentach multiplikowanych, gdzie wiele próbek jest połączonych razem.
Podstawowym mechanizmem stojącym za zmianą indeksów jest związany z chemią i przepływem pracy platform sekwencjonowania, szczególnie tych wykorzystujących wzorowane komórki przepływowe i amplifikację z wykluczeniem, jak niektóre modele od Illumina. Podczas przygotowania bibliotek unikalne sekwencje indeksów są ligowane lub włączane do fragmentów DNA, aby umożliwić identyfikację próbek po sekwencjonowaniu. Jednak w niektórych przypadkach swobodnie unoszące się adaptery lub niekompletne produkty ligacji pozostają w połączonej bibliotece. Podczas generacji klastrów na komórce przepływowej te wolne adaptery mogą przyłączać się do fragmentów DNA z różnych próbek, co skutkuje włączeniem nieprawidłowego indeksu podczas amplifikacji. Proces ten jest zaostrzony w przepływach pracy, które wykorzystują amplifikację z wykluczeniem, gdzie fragmenty DNA są unieruchamiane i amplifikowane w bliskiej odległości, co zwiększa prawdopodobieństwo błędnego przypisania indeksów.
Innym czynnikiem przyczyniającym się jest stosowanie kompozytowych indeksów podwójnych, gdzie dwa indeksy (i5 i i7) są używane w połączeniu, aby zwiększyć zdolność multiplikacji. Jeśli wystąpi zmiana indeksów, odczyt może zostać przypisany do kombinacji indeksów, która nigdy nie była obecna w oryginalnej bibliotece, co utrudnia śledzenie prawdziwego pochodzenia fragmentu. To jest szczególnie problematyczne w aplikacjach wymagających wysokiej czułości, takich jak sekwencjonowanie RNA pojedynczych komórek, gdzie nawet niskie poziomy zmiany indeksów mogą wprowadzać znaczące artefakty.
Wskaźnik zmiany indeksów może być wpływany przez kilka czynników, w tym jakość przygotowania bibliotek, obecność nadmiaru adapterów, używaną platformę sekwencjonowania oraz specyficzną chemię komórki przepływowej. Na przykład, wzorowane komórki przepływowe, które są zaprojektowane, aby zwiększyć gęstość klastrów i przepustowość, były kojarzone z wyższymi wskaźnikami zmiany indeksów w porównaniu do nie-wzorowanych komórek przepływowych. Dodatkowo, stosowanie unikalnych indeksów podwójnych (gdzie każda próbka jest przypisana do unikalnej pary indeksów) może pomóc w złagodzeniu skutków zmiany indeksów, ułatwiając identyfikację i filtrowanie błędnych odczytów.
Zrozumienie mechanizmów stojących za zmianą indeksów jest kluczowe dla badaczy i dostawców sekwencjonowania, takich jak Illumina i Thermo Fisher Scientific, ponieważ informuje o opracowywaniu ulepszonych protokołów przygotowania bibliotek i chemii sekwencjonowania. Trwające badania i postępy technologiczne mają na celu zminimalizowanie zmiany indeksów, co z kolei zwiększa dokładność i niezawodność eksperymentów sekwencjonowania DNA o wysokiej przepustowości.
Czynniki technologiczne: platformy sekwencjonowania i protokoły
Zmiana indeksów, znana również jako skakanie indeksów, to zjawisko w sekwencjonowaniu DNA, w którym indeksy próbki (kody kreskowe) są błędnie przypisywane do odczytów sekwencjonowania, co prowadzi do błędnego przypisania sekwencji między multiplikowanymi próbkami. Problem ten jest szczególnie istotny w platformach sekwencjonowania o wysokiej przepustowości, które wykorzystują strategie kodowania kompozytowego, takie jak te opracowane przez Illumina, globalnego lidera technologii sekwencjonowania nowej generacji (NGS). Czynniki technologiczne przyczyniające się do zmiany indeksów są ściśle związane z projektem platform sekwencjonowania oraz protokołami stosowanymi podczas przygotowania bibliotek i sekwencjonowania.
Architektura platform sekwencjonowania odgrywa kluczową rolę w częstości występowania zmiany indeksów. Na przykład wzorowane komórki przepływowe, które są am używane w zaawansowanych sekwencerach Illumina, takich jak seria NovaSeq, zostały powiązane z wyższymi wskaźnikami skakania indeksów w porównaniu do wcześniejszych, nie-wzorowanych projektów komórek przepływowych. Wynika to częściowo z fizycznej bliskości klastrów DNA i wykorzystania amplifikacji z wykluczeniem, co może ułatwiać transfer swobodnie unoszących się adapterów lub indeksów między klastrami podczas procesu sekwencjonowania. Chemia reakcji sekwencjonowania, w tym użycie określonych polimeraz i obecność nadmiaru adapterów, mogą jeszcze bardziej zaostrzać ten efekt.
Protokóły przygotowania bibliotek są kolejnym istotnym czynnikiem technologicznym. Strategie podwójnego indeksowania, w której oba końce fragmentu DNA są oznaczone unikalnymi indeksami, wykazały, że zmniejszają wpływ zmiany indeksów w porównaniu do metod pojedynczego indeksowania. Jednak nawet przy podwójnym indeksowaniu, niepełne usunięcie swobodnych adapterów lub niewłaściwe kroki czyszczenia mogą pozostawiać resztkowe indeksy w mieszaninie reakcyjnej, zwiększając ryzyko błędnego przypisania. Wybór odczynników, wydajność reakcji enzymatycznych i surowość kroków oczyszczania wpływają na prawdopodobieństwo zdarzeń zmiany indeksów.
Przepustowość sekwencjonowania i poziomy multiplikacji również wpływają na wskaźniki zmiany indeksów. W miarę jak liczba próbek połączonych w jednym cyklu sekwencjonowania wzrasta, prawdopodobieństwo błędnego przypisania indeksu rośnie, szczególnie jeśli indeksy nie są wystarczająco unikalne lub jeśli występuje kontaminacja między próbkami podczas ich obsługi. To jest szczególnie niepokojące w dużych projektach genomowych i zastosowaniach klinicznych, w których dokładna identyfikacja próbek jest kluczowa.
Aby sprostać tym wyzwaniom, producenci platform, tacy jak Illumina i konsorcja badawcze, opracowały najlepsze praktyki, w tym stosowanie unikalnych indeksów podwójnych, rygorystycznych protokołów czyszczenia bibliotek oraz metod obliczeniowych do wykrywania i korygowania artefaktów zmiany indeksów. Spodziewane są dalsze innowacje technologiczne w chemii sekwencjonowania, projektowaniu komórek przepływowych i automatyzacji, które mają na celu zmniejszenie wpływu zmiany indeksów w 2025 roku i później.
Konieczności dla dokładności danych genomicznych i badań
Zmiana indeksów, znana również jako skakanie indeksów, to zjawisko w sekwencjonowaniu DNA o wysokiej przepustowości, w którym indeksy próbki (kody kreskowe) są błędnie przypisywane do odczytów sekwencjonowania. To błędne przypisanie może mieć znaczące konsekwencje dla dokładności danych genomicznych i integralności dalszych badań. W miarę jak platformy sekwencjonowania, szczególnie te wykorzystujące wzorowane komórki przepływowe i określone chemie przygotowania bibliotek, stały się bardziej powszechne, ryzyko i wpływ zmiany indeksów zyskały większą uwagę społeczności genomowej.
Jedną z podstawowych konsekwencji zmiany indeksów jest wprowadzenie kontaminacji między próbkami. Gdy odczyty są błędnie przypisywane do niewłaściwej próbki, może to prowadzić do fałszywych pozytywów – wykrywania wariantów genetycznych lub sekwencji, które nie są rzeczywiście obecne w danej próbce. Jest to szczególnie problematyczne w badaniach dotyczących rzadkich wariantów, wykrywania rzadkich patogenów lub sekwencjonowania pojedynczych komórek, gdzie nawet niewielka liczba błędnie przypisanych odczytów może zaburzać wyniki i prowadzić do błędnych wniosków biologicznych. Na przykład w genomice nowotworowej zmiana indeksów może skutkować błędnym identyfikowaniem mutacji somatycznych, co może wpływać na decyzje diagnostyczne lub terapeutyczne.
Wpływ zmiany indeksów obejmuje również szeroko zakrojone badania populacyjne i metagenomikę, gdzie dokładne demultipleksowanie próbek jest niezbędne do wiarygodnej interpretacji danych. W badań metagenomowych zmiana indeksów może sztucznie zwiększać różnorodność społeczności mikrobiologicznych lub zaciemniać prawdziwe sygnały biologiczne, co utrudnia zrozumienie złożonych ekosystemów. Podobnie, w genetyce populacyjnej błędne przypisanie odczytów może zakłócać analizy struktury genetycznej, przodków i badań asocjacyjnych, podważając ważność wyników badań.
Aby sprostać tym wyzwaniom, dostawcy technologii sekwencjonowania, tacy jak Illumina, opracowali ulepszone protokoły przygotowania bibliotek i strategie podwójnego indeksowania, aby złagodzić ryzyko zmiany indeksów. Podwójne indeksowanie, w którym stosowane są dwa unikalne kody kreskowe na próbkę, znacznie zmniejsza prawdopodobieństwo błędnego przypisania, ponieważ oba indeksy musiałyby się jednocześnie zmienić, aby doszło do błędu. Dodatkowo, narzędzia bioinformatyczne i środki kontroli jakości są coraz częściej wykorzystywane do wykrywania i filtrowania potencjalnie zmienionych odczytów indeksów, choć podejścia te mogą nie całkowicie wyeliminować problem.
Konsekwencje zmiany indeksów podkreślają znaczenie rygorystycznego projektowania eksperymentalnego, starannego wyboru platform sekwencjonowania oraz wdrażania solidnych pipeline’ów analizy danych. Wraz z dalszymi postępami w dziedzinie genomiki, trwałe wysiłki ze strony organizacji takich jak Narodowe Instytuty Zdrowia i Narodowy Instytut Badań Genomu Ludzkiego mają na celu ustalenie najlepszych praktyk i standardów, aby zapewnić dokładność i powtarzalność badań genomicznych w obliczu technicznych wyzwań, takich jak zmiana indeksów.
Metody wykrywania: identyfikacja i kwantyfikacja zmiany indeksów
Zmiana indeksów, znana również jako skakanie indeksów, to zjawisko w multiplikowanym sekwencjonowaniu DNA, gdzie indeksy próbek (kody kreskowe) są błędnie przypisywane do odczytów sekwencjonowania, prowadząc do błędnego przypisania danych między próbkami. Dokładne wykrywanie i kwantyfikacja zmiany indeksów są krytyczne dla zapewnienia integralności danych, szczególnie w zastosowaniach takich jak metagenomika, sekwencjonowanie pojedynczych komórek i diagnostyka kliniczna. Opracowano kilka metod wykrywania w celu identyfikacji i kwantyfikacji zdarzeń zmiany indeksów, wykorzystując zarówno projekt eksperymentalny, jak i analizę obliczeniową.
Podstawowym podejściem do wykrywania zmiany indeksów jest zastosowanie kontroli negatywnych i syntetycznych wstawek. Włączając próbki z unikalnymi, znanymi sekwencjami lub syntetyczne DNA, które nie powinny pokrywać się z próbkami biologicznymi, badacze mogą monitorować obecność nieoczekiwanych kombinacji indeksów. Wykrycie tych nieoczekiwanych kombinacji w danych sekwencjonowania dostarcza bezpośrednich dowodów na wystąpienie zmiany indeksów. Metoda ta jest szeroko zalecana przez dostawców platform sekwencjonowania, takich jak Illumina, wiodącego producenta instrumentów do sekwencjonowania nowej generacji (NGS), które opublikowało wytyczne dotyczące projektowania eksperymentalnego w celu minimalizacji i wykrywania skakania indeksów.
Inną powszechną strategią jest wykorzystanie schematów podwójnego indeksowania, gdzie każda próbka jest oznaczona dwoma unikalnymi indeksami (i5 i i7). Podejście to umożliwia identyfikację zmiany indeksów poprzez wykrywanie par indeksów, które nie były używane podczas przygotowania bibliotek. Narzędzia obliczeniowe mogą następnie kwantyfikować częstotliwość tych nieoczekiwanych par indeksów, dostarczając oszacowania wskaźnika zmiany indeksów. Podwójne indeksowanie jest obecnie standardową praktyką w wielu przepływach pracy sekwencjonowania, jak zalecają organizacje takie jak Illumina oraz Thermo Fisher Scientific, które są głównymi dostawcami odczynników i platform sekwencjonowania.
Analiza bioinformatyczna odgrywa kluczową rolę w wykrywaniu i kwantyfikacji zmiany indeksów. Algorytmy mogą przeszukiwać dane sekwencjonowania w poszukiwaniu odczytów z kombinacjami indeksów, które nie pasują do żadnych oczekiwanych przypisań próbek. Porównując zaobserwowany rozkład par indeksów z oczekiwanym rozkładem, badacze mogą oszacować wskaźnik i wzór zmiany indeksów. Niektóre pipeline’y włączają również modele statystyczne, aby odróżnić prawdziwą zmianę indeksów od błędów sekwencjonowania lub kontaminacji. Narodowe Instytuty Zdrowia (NIH), główny agencja badawcza w dziedzinie biomedycyny, wspiera rozwój narzędzi open-source i najlepszych praktyk do analizy danych sekwencjonowania multiplikowanego, podkreślając znaczenie solidnych metod detekcji obliczeniowej.
Podsumowując, wykrywanie i kwantyfikacja zmiany indeksów w sekwencjonowaniu DNA polegają na połączeniu kontroli eksperymentalnych, strategii podwójnego indeksowania oraz zaawansowanych analiz bioinformatycznych. Przestrzeganie najlepszych praktyk zalecanych przez wiodące organizacje i dostawców technologii sekwencjonowania jest niezbędne do minimalizacji wpływu zmiany indeksów i zapewnienia niezawodności wyników sekwencjonowania.
Strategie łagodzenia: rozwiązania laboratoryjne i bioinformatyczne
Zmiana indeksów, znana również jako skakanie indeksów, to dobrze udokumentowany artefakt w sekwencjonowaniu DNA o wysokiej przepustowości, szczególnie w eksperymentach multiplikowanych, gdzie wiele próbek jest połączonych i odróżnionych przez unikalne sekwencje indeksów. To zjawisko może prowadzić do błędnego przypisania odczytów, kompromitując integralność danych i dalsze analizy. W miarę jak technologie i zastosowania sekwencjonowania rozwijają się w 2025 roku, solidne strategie łagodzenia – zarówno na poziomie laboratoryjnym, jak i bioinformatycznym – są niezbędne, aby zapewnić dokładność danych.
Rozwiązania laboratoryjne
- Indeksowanie podwójne: Jedną z najbardziej efektywnych strategii laboratoryjnych jest zastosowanie unikalnych indeksów podwójnych (UDI), gdzie każda próbka jest oznaczona dwiema odrębnymi sekwencjami indeksów. Podejście to znacznie zmniejsza prawdopodobieństwo błędnego przypisania, ponieważ oba indeksy musiałyby się jednocześnie zmienić, aby odczyt został błędnie przypisany. Główni dostawcy platform sekwencjonowania, tacy jak Illumina, wprowadzili zestawy i protokoły UDI, aby zająć się tym problemem.
- Optymalizacja przygotowania bibliotek: Staranna optymalizacja protokołów przygotowania bibliotek może zminimalizować kontaminację swobodnymi adapterami, która jest znanym czynnikiem przyczyniającym się do zmiany indeksów. Obejmuje to dokładne czyszczenie oparte na kulkach i enzymatyczne usuwanie nadmiaru adapterów. Organizacje takie jak Thermo Fisher Scientific dostarczają wytyczne i odczynniki wspierające te najlepsze praktyki.
- Wybór platformy i aktualizacje chemii: Niektóre platformy sekwencjonowania i chemie są bardziej podatne na zmiany indeksów niż inne. Na przykład, wzorowane komórki przepływowe i technologie amplifikacji z wykluczeniem były związane z wyższymi wskaźnikami skakania indeksów. Bycie na bieżąco z najnowszymi ulepszonymi platformami i wydaniami chemicznymi od producentów może pomóc laboratoriom w wyborze systemów o niższych wskaźnikach zmiany indeksów.
Rozwiązania bioinformatyczne
- Rygorystyczne algorytmy demultipleksowania: Zaawansowane narzędzia do demultipleksowania mogą być konfigurowane, aby wymagać doskonałych dopasowań do obu sekwencji indeksów, odrzucając odczyty z niejednoznacznymi lub nieoczekiwanymi kombinacjami indeksów. To zmniejsza ryzyko błędnie przypisanych odczytów, które trafiają do dalszych analiz.
- Statystyczne filtrowanie i wykrywanie kontaminacji: Pipeline’y bioinformatyczne mogą włączać modele statystyczne do identyfikacji i filtrowania rzadkich kombinacji indeksów, które prawdopodobnie są wynikiem zmiany indeksów. Niektóre pipeline’y również oznaczają lub usuwają odczyty, które pojawiają się w niespodziewanych parach indeksów, co dodatkowo poprawia jakość danych.
- Ocena kontaminacji między próbkami: Regularna ocena kontaminacji między próbkami z użyciem kontroli wewnętrznych lub syntetycznych wstawek może pomóc w kwantyfikacji i korekcji artefaktów zmiany indeksów. To jest szczególnie ważne w wrażliwych zastosowaniach, takich jak sekwencjonowanie pojedynczych komórek lub wykrywanie rzadkich wariantów.
Podsumowując, połączenie najlepszych praktyk laboratoryjnych i zaawansowanych podejść bioinformatycznych jest wymagane, aby złagodzić zmianę indeksów w sekwencjonowaniu DNA. Trwała współpraca między dostawcami technologii sekwencjonowania, takimi jak Illumina i Thermo Fisher Scientific, oraz społecznością naukową wciąż napędza poprawę zarówno projektowania eksperymentalnego, jak i analizy danych, zapewniając niezawodność danych sekwencjonowania multiplikowanego w 2025 roku i w przyszłości.
Standardy branżowe i wytyczne (np. Illumina, NIH)
Zmiana indeksów, znana również jako skakanie indeksów, to dobrze rozpoznawany artefakt techniczny w sekwencjonowaniu DNA o wysokiej przepustowości, szczególnie w przepływach sekwencjonowania multiplikowanego. To zjawisko występuje, gdy indeksy próbek (kody kreskowe) są błędnie przypisywane do odczytów sekwencjonowania, prowadząc do błędnego przypisania danych między próbkami. W miarę jak adopcja sekwencjonowania nowej generacji (NGS) rozszerza się w badaniach, diagnostyce klinicznej i zastosowaniach przemysłowych, potrzeba solidnych standardów branżowych i wytycznych do łagodzenia i monitorowania zmiany indeksów staje się coraz bardziej krytyczna.
Główni dostawcy platform sekwencjonowania, tacy jak Illumina, odegrali kluczową rolę w ustanawianiu najlepszych praktyk dotyczących minimalizacji zmiany indeksów. Illumina, globalny lider technologii NGS, opublikowała notatki techniczne i protokoły dotyczące przyczyn zmiany indeksów, która jest szczególnie powszechna w wzorowanych platformach komórkowych i w przypadku używania bibliotek z pojedynczym indeksowaniem. Ich rekomendacje obejmują stosowanie unikalnych strategii podwójnego indeksowania (UDI), które stosują dwa niezależne kody kreskowe na próbkę, znacznie zmniejszając ryzyko błędnego przypisania. Illumina dostarcza również zwalidowane zestawy indeksów oraz narzędzia oprogramowania do demultipleksowania, które są zaprojektowane do wykrywania i korygowania potencjalnych zdarzeń zmiany indeksów.
Oprócz wytycznych producentów, szersze organizacje naukowe i regulacyjne przyczyniły się do rozwoju standardów. Narodowe Instytuty Zdrowia (NIH), jako wiodąca agencja badawcza biomedycyny w Stanach Zjednoczonych, wydały wskazówki dla badaczy korzystających z NGS w projektach finansowanych przez rząd. NIH zachęca do przyjęcia strategii podwójnego indeksowania i rygorystycznych działań kontrolnych, zwłaszcza w badaniach, gdzie kontaminacja między próbkami mogłaby prowadzić do kompromitacji integralności danych lub bezpieczeństwa pacjentów. Te rekomendacje są często uwzględniane w wymaganiach dotyczących dotacji i politykach udostępniania danych.
Na całym świecie organizacje takie jak Międzynarodowa Organizacja Normalizacyjna (ISO) opracowują standardy dla praktyk laboratoryjnych w genomice, w tym ISO 20387 dla biobanków i ISO 15189 dla laboratoriów medycznych. Choć nie zawsze specyficzne dla zmiany indeksów, te standardy podkreślają ścisłe śledzenie, walidację metod i dokumentację – zasady, które leżą u podstaw skutecznego wykrywania i łagodzenia błędnego przypisania indeksów.
Ponadto, stowarzyszenia zawodowe i konsorcja, w tym Global Alliance for Genomics and Health (GA4GH), opublikowały ramy najlepszych praktyk dotyczących jakości danych NGS i śledzenia próbek. Te ramy często odnoszą się do protokołów producentów i wytycznych regulacyjnych, promując harmonizację wśród laboratoriów i jurysdykcji.
Podsumowując, standardy branżowe i wytyczne dotyczące rozwiązywania zmiany indeksów w sekwencjonowaniu DNA kształtowane są przez połączenie protokołów producentów, zaleceń krajowych agencji badawczych oraz międzynarodowych standardów laboratoryjnych. Przestrzeganie tych wytycznych jest istotne dla zapewnienia dokładności danych, powtarzalności oraz wiarygodności dalszych analiz zarówno w badaniach, jak i w kontekście klinicznym.
Rynek i zainteresowanie opinii publicznej: aktualne trendy i prognozy na 5 lat
Zmiana indeksów, znana również jako skakanie indeksów, to zjawisko w sekwencjonowaniu DNA, gdzie indeksy próbek (kody kreskowe) są błędnie przypisywane do odczytów sekwencjonowania, prowadząc do kontaminacji między multiplikowanymi próbkami. Problem ten jest szczególnie istotny w platformach sekwencjonowania o wysokiej przepustowości, takich jak te opracowane przez Illumina, globalnego lidera technologii genomowych. W miarę jak adopcja sekwencjonowania nowej generacji (NGS) rozszerza się w diagnostyce klinicznej, badaniach i biotechnologii, rynek oraz zainteresowanie opinii publicznej w rozwiązywaniu zmiany indeksów znacznie wzrosły.
W 2025 roku rynek sekwencjonowania DNA nadal doświadcza dynamicznego wzrostu, napędzanego rosnącym zapotrzebowaniem na medycynę precyzyjną, genomikę populacyjną i nadzór nad chorobami zakaźnymi. Globalny rynek NGS ma szansę na wzrost o ponad 15% CAGR, a Ameryka Północna, Europa i region Azji-Pacyfiku są kluczowymi obszarami działalności. W tym kontekście integralność danych sekwencjonowania jest kluczowa, a zmiana indeksów stała się istotną kwestią jakościową. Główni dostawcy platform sekwencjonowania, w tym Illumina i Thermo Fisher Scientific, reagowali, opracowując ulepszone zestawy do przygotowania bibliotek, strategie podwójnego indeksowania oraz rozwiązania programowe w celu zmniejszenia ryzyka błędnego przypisania indeksów.
Zainteresowanie opinii publicznej wiarygodnością danych sekwencjonowania również rośnie, szczególnie w miarę jak informacje genomowe stają się nieodłącznym elementem decyzji zdrowotnych i polityki zdrowia publicznego. Ciała regulacyjne, takie jak amerykańska Administracja Żywności i Leków (FDA) oraz międzynarodowe organizacje takie jak Światowa Organizacja Zdrowia zwracają coraz większą uwagę na standardy i powtarzalność analiz genomowych, w tym wpływ artefaktów technicznych, takich jak zmiana indeksów. Doprowadziło to do publikacji wytycznych najlepszych praktyk i włączenia metryk jakości do procesów sekwencjonowania klinicznego.
Patrząc w przyszłość na następne pięć lat, trend zmierza w kierunku większej automatyzacji, wyższej przepustowości i bardziej złożonego multiplikowania w przepływach pracy sekwencjonowania. Prawdopodobnie to zwiększy potencjał zmiany indeksów, chyba że przeciwdziałać temu będą kontynuowane innowacje. Oczekuje się, że rynek zobaczy dalsze inwestycje w solidne chemie indeksujące, algorytmy korekcji błędów i usługi weryfikacji stron trzecich. Dodatkowo, w miarę jak sekwencjonowanie jest przyjmowane w zdecentralizowanych i punktowych kontekstach opieki zdrowotnej, na czoło pojawią się przyjazne użytkownikowi rozwiązania do minimalizowania zmiany indeksów.
Podsumowując, rynek i zainteresowanie opinii publicznej w zakresie zmiany indeksów w sekwencjonowaniu DNA mają intensyfikować się do 2030 roku, napędzane rosnącą rolą genomiki w medycynie i badaniach. Interesariusze – w tym deweloperzy technologii, agencje regulacyjne i użytkownicy końcowi – będą priorytetować rozwiązania, które zapewniają integralność danych, wspierając dalszy rozwój i zaufanie do aplikacji opartych na sekwencjonowaniu.
Przyszłe spojrzenie: innowacje, wyzwania i droga naprzód
Zmiana indeksów, znana również jako skakanie indeksów, pozostaje istotnym problemem w sekwencjonowaniu DNA o wysokiej przepustowości, szczególnie w eksperymentach multiplikowanych, gdzie próbki są łączone i odróżniane przez unikalne sekwencje indeksów. W miarę jak technologie sekwencjonowania się rozwijają, a zastosowania stają się coraz bardziej zróżnicowane – od diagnostyki klinicznej po szeroko zakrojone badania populacyjne – potrzeba zajęcia się zmianą indeksów staje się coraz bardziej pilna. Patrząc w przyszłość na 2025 roku, przyszłe spojrzenie na zarządzanie i łagodzenie zmiany indeksów kształtuje się zarówno przez innowacje technologiczne, jak i utrzymujące się wyzwania.
Jednym z najbardziej obiecujących obszarów innowacji jest rozwój ulepszonych chemii przygotowania bibliotek oraz platform sekwencjonowania. Główni dostawcy technologii sekwencjonowania, tacy jak Illumina i Thermo Fisher Scientific, aktywnie doskonalą swoje odczynniki i protokoły, aby zminimalizować ryzyko błędnego przypisania indeksów. Na przykład, przyjęcie unikalnych strategii podwójnego indeksowania (UDI) – gdzie używane są dwie niezależne sekwencje indeksów na próbkę – już wykazało znaczne zmniejszenie wydarzeń zmiany indeksów. Oczekuje się, że dalsze ulepszenia w syntezie i oczyszczaniu oligonukleotydów obniżą poziom szumów tła, które przyczyniają się do błędnego przypisania.
Na froncie obliczeniowym narzędzia bioinformatyczne ewoluują, aby lepiej wykrywać i korygować zmiany indeksów. Algorytmy modelujące oczekiwany rozkład kombinacji indeksów i oznaczające anomalne wzorce są integrowane z standardowymi procesami analizy danych sekwencjonowania. Te postępy są wspierane przez współpracę organizacji takich jak Narodowe Instytuty Zdrowia (NIH), która finansuje badania dotyczące zarówno rozwiązań eksperymentalnych, jak i obliczeniowych dotyczących artefaktów sekwencjonowania.
Pomimo tych postępów, wiele wyzwań pozostaje. W miarę wzrostu przepustowości sekwencjonowania i coraz powszechniejszego stosowania multiplikacji próbek, nawet niskie wskaźniki zmiany indeksów mogą znacząco wpłynąć na jakość danych, szczególnie w zastosowaniach wymagających wysokiej czułości, takich jak wykrywanie rzadkich wariantów czy sekwencjonowanie pojedynczych komórek. Dodatkowo, różnorodność platform sekwencjonowania i chemii komplikuje rozwój uniwersalnych rozwiązań. Współpraca między organizacjami, takimi jak Narodowy Instytut Badań Genomu Ludzkiego (NHGRI), w celu standaryzacji najlepszych praktyk w branży będzie kluczowa do zapewnienia integralności danych.
Patrząc w przyszłość, droga do minimalizacji zmiany indeksów prawdopodobnie będzie obejmować połączenie ulepszonych protokołów laboratoryjnych, solidnych metod korekcji obliczeniowej oraz standardów w całej branży. Ciągła współpraca między deweloperami technologii, instytucjami badawczymi i agencjami regulacyjnymi będzie niezbędna, aby zapewnić, że korzyści z sekwencjonowania o wysokiej przepustowości nie będą podważane przez artefakty techniczne. W miarę jak dziedzina zmierza ku coraz większym i bardziej złożonym projektom sekwencjonowania, zajmowanie się zmianą indeksów pozostanie priorytetem dla społeczności genomowej.
Źródła i odniesienia
- Narodowe Instytuty Zdrowia
- Illumina
- Thermo Fisher Scientific
- Thermo Fisher Scientific
- Narodowe Instytuty Zdrowia
- Międzynarodowa Organizacja Normalizacyjna
- Światowa Organizacja Zdrowia
https://youtube.com/watch?v=WKAUtJQ69n8