Changement d’index dans le séquençage ADN : La menace silencieuse pour l’intégrité des données et comment les technologies de pointe s’attaquent à ce problème. Découvrez l’impact, la détection et l’avenir de ce phénomène critique. (2025)
- Introduction : Qu’est-ce que le changement d’index dans le séquençage ADN ?
- Contexte historique et découverte du changement d’index
- Mécanismes derrière le changement d’index : Comment et pourquoi cela se produit
- Facteurs technologiques : Plateformes de séquençage et protocoles
- Conséquences pour la précision des données génomiques et la recherche
- Méthodes de détection : Identifier et quantifier le changement d’index
- Stratégies d’atténuation : Solutions de laboratoire et de bioinformatique
- Normes et directives de l’industrie (ex. : Illumina, NIH)
- Marché et intérêt public : Tendances actuelles et prévisions sur 5 ans
- Perspectives d’avenir : Innovations, défis et voie à suivre
- Sources & Références
Introduction : Qu’est-ce que le changement d’index dans le séquençage ADN ?
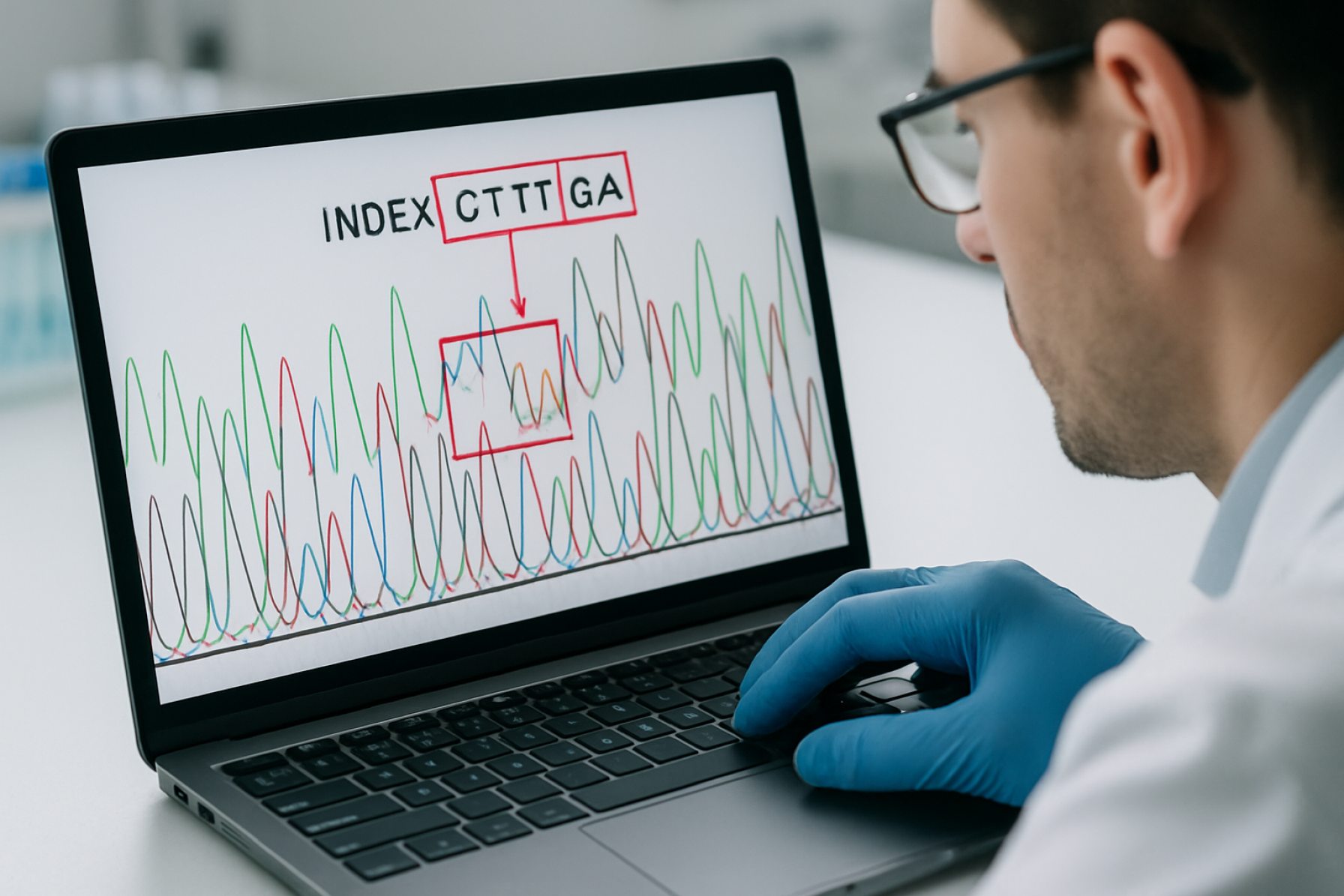
Le changement d’index, également connu sous le nom de « saut d’index », est un phénomène observé sur les plates-formes de séquençage ADN à haut débit, en particulier celles utilisant des stratégies de séquençage multiplexé. Dans ces approches, des séquences d’ADN courtes et uniques—appelées index ou codes-barres—sont attachées à des échantillons ADN individuels. Cela permet de regrouper plusieurs échantillons et de les séquencer ensemble en une seule course, avec l’attente que chaque lecture puisse ensuite être attribuée avec précision à son échantillon d’origine en fonction de son index. Cependant, le changement d’index se produit lorsque la séquence d’index associée à un fragment d’ADN est incorrectement attribuée, entraînant des lectures attribuées à tort à l’échantillon incorrect.
Cette mauvaise attribution peut survenir à différentes étapes du flux de travail de séquençage, y compris la préparation de la bibliothèque, la génération de clusters et le séquençage lui-même. Le problème est particulièrement prononcé sur les plates-formes utilisant des cellules d’écoulement à motifs et une amplification d’exclusion, telles que celles développées par Illumina, Inc., un fournisseur de technologie de séquençage de nouvelle génération (NGS) de premier plan. Dans ces systèmes, des adaptateurs flottants ou des événements de ligature incomplets peuvent entraîner le transfert de séquences d’index entre les fragments d’ADN, provoquant ainsi une sous-ensemble de lectures portant le mauvais index.
Les conséquences du changement d’index sont significatives pour de nombreuses applications de séquençage ADN. Dans les études où des variantes rares ou des signaux de faible abondance sont importants—comme en génomique unicellulaire, en métagénomique ou en diagnostics cliniques—même un faible taux de mauvaise attribution d’index peut conduire à des faux positifs, à de la contamination ou à des conclusions erronées. À mesure que le débit de séquençage et les niveaux de multiplexage augmentent, l’impact potentiel du changement d’index sur la qualité des données et leur interprétation devient plus prononcé.
La prise de conscience du changement d’index a poussé la communauté génomique à développer des stratégies expérimentales et computationnelles pour atténuer ses effets. Cela inclut l’utilisation d’index doubles uniques (UDI), de protocoles de préparation de bibliothèque améliorés et de méthodes de filtrage bioinformatique. Des organisations telles que les National Institutes of Health (NIH) et le National Human Genome Research Institute (NHGRI) ont souligné l’importance de l’identification précise des échantillons dans les études de séquençage, soulignant la nécessité de solutions robustes au changement d’index.
En résumé, le changement d’index est un défi technique critique dans le séquençage ADN moderne, avec des implications pour l’intégrité des données, la reproductibilité et la fiabilité des résultats scientifiques et cliniques. Comprendre ses mécanismes et développer des contre-mesures efficaces restent des domaines de recherche et d’innovation actifs dans le domaine de la génomique.
Contexte historique et découverte du changement d’index
Le phénomène de changement d’index, également connu sous le nom de « saut d’index », est devenu une préoccupation importante dans le domaine du séquençage ADN à haut débit au milieu des années 2010. Le changement d’index se réfère à la mauvaise attribution des indices d’échantillon (codes-barres) pendant les courses de séquençage multiplexées, résultant en lectures attribuées incorrectement à l’échantillon erroné. Cet artefact peut compromettre la précision des analyses ultérieures, en particulier dans les applications nécessitant une haute sensibilité, telles que la génomique unicellulaire et la métagénomique.
Le contexte historique du changement d’index est étroitement lié à l’évolution rapide des technologies de séquençage de nouvelle génération (NGS). Les premières plates-formes NGS, telles que celles développées par Illumina et Thermo Fisher Scientific, ont permis le séquençage simultané de plusieurs échantillons en attachant des séquences d’index uniques à chaque bibliothèque. Cette approche de multiplexage a considérablement augmenté le débit et réduit les coûts, mais a également introduit de nouvelles sources d’erreur. Au départ, l’accent était mis sur la minimisation de la contamination croisée lors de la préparation des bibliothèques et du séquençage. Cependant, à mesure que la profondeur de séquençage et la sensibilité s’amélioraient, les chercheurs ont commencé à observer des motifs inattendus de mauvaise attribution des lectures qui ne pouvaient pas être expliqués par une contamination traditionnelle.
La découverte du changement d’index en tant qu’artefact technique distinct a été systématiquement décrite pour la première fois en 2017, lorsque des études utilisant les plates-formes de cellules d’écoulement à motifs d’Illumina (telles que HiSeq 4000 et NovaSeq) ont signalé des taux élevés de mauvaise attribution d’index. Les chercheurs ont constaté que l’utilisation de la chimie d’amplification d’exclusion (ExAmp), qui a remplacé l’amplification en pont dans ces nouvelles plates-formes, était associée à une augmentation des événements de changement d’index. Cela était attribué à la présence d’adaptateurs flottants et à la proximité physique des clusters sur les cellules d’écoulement à motifs, facilitant le transfert de séquences d’index entre les bibliothèques lors de la génération de clusters. Le problème était particulièrement prononcé dans les expériences de RNA-seq unicellulaire, où même de faibles niveaux de changement d’index pouvaient conduire à des artefacts de données significatifs.
En réponse à ces résultats, des fournisseurs de technologie de séquençage tels qu’Illumina ont reconnu le problème et ont commencé à recommander des meilleures pratiques pour atténuer le changement d’index, y compris l’utilisation d’index doubles uniques et d’améliorations des protocoles de nettoyage des bibliothèques. La communauté génomique au sens large, y compris des organisations comme le National Human Genome Research Institute (NHGRI), a depuis souligné l’importance de comprendre et de contrôler le changement d’index dans la conception expérimentale et l’interprétation des données. À partir de 2025, des recherches continues visent à affiner les chimies de séquençage et les approches bioinformatiques pour réduire encore l’impact du changement d’index sur les études génomiques.
Mécanismes derrière le changement d’index : Comment et pourquoi cela se produit
Le changement d’index, également connu sous le nom de saut d’index, est un phénomène dans le séquençage ADN à haut débit où les séquences d’index spécifiques à l’échantillon (codes-barres) attribuées lors de la préparation de la bibliothèque sont incorrectement associées à des fragments d’ADN d’autres échantillons. Cette mauvaise attribution peut entraîner une contamination croisée des lectures de séquençage, pouvant confondre les analyses ultérieures, en particulier dans les expériences multiplexées où de nombreux échantillons sont regroupés.
Le principal mécanisme derrière le changement d’index est lié à la chimie et au flux de travail des plates-formes de séquençage, en particulier celles utilisant des cellules d’écoulement à motifs et une amplification d’exclusion, comme certains modèles d’Illumina. Lors de la préparation de la bibliothèque, des séquences d’index uniques sont ligaturées ou incorporées dans des fragments d’ADN pour permettre l’identification des échantillons après le séquençage. Cependant, dans certains cas, des adaptateurs flottants ou des produits de ligature incomplets restent dans la bibliothèque poolée. Lors de la génération de clusters sur la cellule d’écoulement, ces adaptateurs libres peuvent s’assembler à des fragments d’ADN d’échantillons différents, entraînant l’incorporation d’un index incorrect lors de l’amplification. Ce processus est exacerbé dans les flux de travail utilisant l’amplification d’exclusion, où les fragments d’ADN sont immobilisés et amplifiés à proximité, augmentant la probabilité de mauvaise attribution d’index.
Un autre facteur contribuant est l’utilisation de l’indexation binaire combinatoire, où deux indices (i5 et i7) sont utilisés en combinaison pour augmenter la capacité de multiplexage. Si un changement d’index se produit, une lecture peut se voir attribuer une combinaison d’indices qui n’était jamais présente dans la bibliothèque d’origine, rendant difficile la traçabilité de la véritable origine du fragment. C’est particulièrement problématique dans les applications nécessitant une haute sensibilité, telle que le séquençage RNA unicellulaire, où même de faibles niveaux de changement d’index peuvent introduire des artefacts importants.
Le taux de changement d’index peut être influencé par plusieurs facteurs, y compris la qualité de la préparation de la bibliothèque, la présence d’adaptateurs excédentaires, la plate-forme de séquençage utilisée et la chimie spécifique de la cellule d’écoulement. Par exemple, les cellules d’écoulement à motifs, conçues pour augmenter la densité de clusters et le débit, ont été associées à des taux de changement d’index plus élevés par rapport aux cellules d’écoulement non à motifs. De plus, l’utilisation d’index doubles uniques (où chaque échantillon se voit attribuer une paire d’index unique) peut aider à atténuer les effets du changement d’index en facilitant l’identification et le filtrage des lectures mal attribuées.
Comprendre les mécanismes derrière le changement d’index est crucial pour les chercheurs et les fournisseurs de séquençage, comme Illumina et Thermo Fisher Scientific, car cela informe le développement de protocoles de préparation de bibliothèque améliorés et de chimies de séquençage. Des recherches en cours et des avancées technologiques visent à minimiser le changement d’index, renforçant ainsi la précision et la fiabilité des expériences de séquençage ADN multiplexé.
Facteurs technologiques : Plateformes de séquençage et protocoles
Le changement d’index, également connu sous le nom de saut d’index, est un phénomène dans le séquençage ADN où les indices d’échantillon (codes-barres) sont incorrectement attribués aux lectures de séquençage, conduisant à une mauvaise attribution de séquences entre des échantillons multiplexés. Ce problème est particulièrement pertinent sur les plates-formes de séquençage à haut débit qui utilisent des stratégies de codage combinatoire, telles que celles développées par Illumina, un leader mondial dans la technologie de séquençage de nouvelle génération (NGS). Les facteurs technologiques contribuant au changement d’index sont étroitement liés à la conception des plates-formes de séquençage et aux protocoles employés lors de la préparation de la bibliothèque et des courses de séquençage.
L’architecture des plates-formes de séquençage joue un rôle critique dans la prévalence du changement d’index. Par exemple, les cellules d’écoulement à motifs, qui sont utilisées dans des séquenceurs avancés d’Illumina comme la série NovaSeq, ont été associées à des taux plus élevés de saut d’index par rapport à des conceptions de cellules d’écoulement non à motifs antérieures. Cela est en partie dû à la proximité physique des clusters d’ADN et à l’utilisation d’amplification d’exclusion, ce qui peut faciliter le transfert d’adaptateurs ou d’indices flottants entre les clusters pendant le processus de séquençage. La chimie de la réaction de séquençage, y compris l’utilisation de certains polymérases et la présence d’adaptateurs excédentaires, peut également aggraver cet effet.
Les protocoles de préparation de la bibliothèque sont un autre facteur technologique significatif. Les stratégies d’indexation double, où chaque extrémité du fragment d’ADN est marquée avec des index uniques, ont montré qu’elles réduisent l’impact du changement d’index par rapport aux méthodes d’indexation unique. Cependant, même avec une indexation double, l’élimination incomplète des adaptateurs flottants ou des étapes de nettoyage inappropriées peuvent laisser des indices résiduels dans le mélange réactionnel, augmentant le risque de mauvaise attribution. Le choix des réactifs, l’efficacité des réactions enzymatiques et la rigueur des étapes de purification influencent tous la probabilité d’événements de changement d’index.
Le débit du séquençage et les niveaux de multiplexage affectent également les taux de changement d’index. À mesure que le nombre d’échantillons regroupés dans une seule course de séquençage augmente, la probabilité de mauvaise attribution d’index augmente, surtout si les indices ne sont pas suffisamment uniques ou s’il y a une contamination croisée lors de la manipulation des échantillons. C’est une préoccupation particulière dans les projets de génomique à grande échelle et les applications cliniques, où l’identification précise des échantillons est primordiale.
Pour relever ces défis, les fabricants de plates-formes comme Illumina et les consortiums de recherche ont développé des meilleures pratiques, y compris l’utilisation d’index doubles uniques, des protocoles de nettoyage rigoureux des bibliothèques et des méthodes computationnelles pour détecter et corriger les artefacts de changement d’index. Les innovations technologiques continues dans la chimie de séquençage, la conception de cellules d’écoulement et l’automatisation devraient encore atténuer l’impact du changement d’index en 2025 et au-delà.
Conséquences pour la précision des données génomiques et la recherche
Le changement d’index, également connu sous le nom de saut d’index, est un phénomène dans le séquençage ADN à haut débit où les indices d’échantillons (codes-barres) sont incorrectement attribués aux lectures de séquençage. Cette mauvaise attribution peut avoir des conséquences significatives sur la précision des données génomiques et l’intégrité des recherches ultérieures. Alors que les plates-formes de séquençage, notamment celles utilisant des cellules d’écoulement à motifs et certaines chimies de préparation de bibliothèque, sont devenues plus répandues, le risque et l’impact du changement d’index ont suscité une attention accrue de la part de la communauté génomique.
Une des principales conséquences du changement d’index est l’introduction de contamination croisée entre échantillons. Lorsque les lectures sont attribuées incorrectement à l’échantillon erroné, cela peut conduire à des faux positifs—détecter des variantes génétiques ou des séquences qui ne sont pas réellement présentes dans un échantillon donné. Cela est particulièrement problématique dans les études impliquant des variantes à faible fréquence, la détection de pathogènes rares ou le séquençage unicellulaire, où même un petit nombre de lectures mal attribuées peut fausser les résultats et conduire à des conclusions biologiques erronées. Par exemple, en génomique du cancer, le changement d’index peut entraîner une identification erronée des mutations somatiques, affectant potentiellement les décisions diagnostiques ou thérapeutiques.
L’impact du changement d’index s’étend aux études de population à grande échelle et à la métagénomique, où une démultiplexion précise des échantillons est essentielle pour une interprétation fiable des données. Dans les enquêtes métagénomiques, le changement d’index peut faussement gonfler la diversité des communautés microbiennes ou obscurcir les véritables signaux biologiques, compliquant les efforts pour comprendre des écosystèmes complexes. De même, en génétique des populations, l’allocation inappropriée des lectures peut confondre les analyses de la structure génétique, des ancêtres et des études d’association, sapant la validité des résultats de recherche.
Pour relever ces défis, des fournisseurs de technologie de séquençage tels qu’Illumina ont développé des protocoles de préparation de bibliothèque améliorés et des stratégies d’indexation double pour atténuer le risque de changement d’index. L’indexation double, où deux codes-barres uniques sont utilisés par échantillon, réduit considérablement la probabilité de mauvaise attribution, car les deux indices devraient devoir changer simultanément pour qu’une erreur se produise. De plus, des outils bioinformatiques et des mesures de contrôle de qualité sont de plus en plus utilisés pour détecter et filtrer les éventuelles lectures switchées par index, bien que ces approches puissent ne pas éliminer complètement le problème.
Les conséquences du changement d’index soulignent l’importance d’une conception expérimentale rigoureuse, d’une sélection soigneuse des plates-formes de séquençage et de la mise en œuvre de pipelines d’analyse de données robustes. Alors que le domaine de la génomique continue d’avancer, des efforts continus d’organisations telles que les National Institutes of Health et le National Human Genome Research Institute visent à établir des meilleures pratiques et des normes pour garantir l’exactitude et la reproductibilité des recherches génomiques face à des défis techniques tels que le changement d’index.
Méthodes de détection : Identifier et quantifier le changement d’index
Le changement d’index, également connu sous le nom de saut d’index, est un phénomène dans le séquençage ADN multiplexé où les indices d’échantillons (codes-barres) sont incorrectement attribués aux lectures de séquençage, entraînant une mauvaise attribution de données entre les échantillons. La détection et la quantification précises du changement d’index sont essentielles pour garantir l’intégrité des données, en particulier dans des applications telles que la métagénomique, le séquençage unicellulaire et les diagnostics cliniques. Plusieurs méthodes de détection ont été développées pour identifier et quantifier les événements de changement d’index, en tirant parti à la fois de la conception expérimentale et de l’analyse computationnelle.
Une approche fondamentale pour détecter le changement d’index implique l’utilisation de contrôles négatifs et de spike-ins synthétiques. En incluant des échantillons avec des séquences uniques et connues ou de l’ADN synthétique qui ne devrait pas chevaucher des échantillons biologiques, les chercheurs peuvent surveiller la présence de combinaisons d’index inattendues. La détection de ces combinaisons inattendues dans les données de séquençage fournit des preuves directes du changement d’index. Cette méthode est largement recommandée par les fournisseurs de plates-formes de séquençage tels qu’Illumina, un fabricant de premier plan d’instruments de séquençage de nouvelle génération (NGS), qui a publié des lignes directrices pour la conception expérimentale afin de minimiser et détecter le saut d’index.
Une autre stratégie commune est l’utilisation de schémas de double-indexation, où chaque échantillon est étiqueté avec deux indices uniques (i5 et i7). Cette approche permet d’identifier le changement d’index en détectant les paires d’index qui n’ont pas été utilisées lors de la préparation de la bibliothèque. Les outils computationnels peuvent ensuite quantifier la fréquence de ces paires d’index inattendues, fournissant une estimation du taux de changement d’index. L’indexation double est désormais une pratique standard dans de nombreux flux de travail de séquençage, comme recommandé par des organisations telles qu’Illumina et Thermo Fisher Scientific, qui sont tous deux des fournisseurs majeurs de réactifs et de plates-formes de séquençage.
L’analyse bioinformatique joue un rôle crucial dans la détection et la quantification du changement d’index. Des algorithmes peuvent analyser les données de séquençage à la recherche de lectures avec des combinaisons d’index qui ne correspondent à aucune des affectations d’échantillons attendues. En comparant la distribution observée des paires d’index à la distribution attendue, les chercheurs peuvent estimer le taux et le schéma de changement d’index. Certains pipelines incorporent également des modèles statistiques pour distinguer le véritable changement d’index des erreurs de séquençage ou de contamination croisée. Les National Institutes of Health (NIH), un important organisme de recherche biomédicale, a soutenu le développement d’outils open-source et de meilleures pratiques pour analyser les données de séquençage multiplexé, soulignant l’importance de méthodes de détection computationnelles robustes.
En résumé, la détection et la quantification du changement d’index dans le séquençage ADN reposent sur une combinaison de contrôles expérimentaux, de stratégies d’indexation double et d’analyses bioinformatiques avancées. Adhérer aux meilleures pratiques recommandées par les organisations de premier plan et les fournisseurs de technologie de séquençage est essentiel pour minimiser l’impact du changement d’index et garantir la fiabilité des résultats de séquençage.
Stratégies d’atténuation : Solutions de laboratoire et de bioinformatique
Le changement d’index, également connu sous le nom de saut d’index, est un artefact bien documenté dans le séquençage ADN à haut débit, en particulier dans les expériences multiplexées où plusieurs échantillons sont regroupés et distingués par des séquences d’index uniques. Ce phénomène peut conduire à une mauvaise attribution des lectures, compromettant ainsi l’intégrité des données et les analyses ultérieures. Alors que les technologies et les applications de séquençage s’élargissent en 2025, des stratégies d’atténuation robustes—tant au niveau du laboratoire qu’en bioinformatique—sont essentielles pour garantir l’exactitude des données.
Solutions de laboratoire
- Indexation double : L’une des stratégies de laboratoire les plus efficaces est l’utilisation d’index doubles uniques (UDI), où chaque échantillon est étiqueté avec deux séquences d’index distinctes. Cette approche réduit considérablement la probabilité de mauvaise attribution, car les deux indices devraient devoir changer simultanément pour qu’une lecture soit attribuée incorrectement. Les principaux fournisseurs de plates-formes de séquençage, tels qu’Illumina, ont intégré des kits et des protocoles UDI pour remédier à ce problème.
- Préparation optimisée des bibliothèques : L’optimisation minutieuse des protocoles de préparation des bibliothèques peut minimiser la contamination par des adaptateurs libres, un contributeur connu au changement d’index. Cela inclut des nettoyages minutieux basés sur des billes et l’élimination enzymatique des adaptateurs excédentaires. Des organisations telles que Thermo Fisher Scientific fournissent des directives et des réactifs pour soutenir ces meilleures pratiques.
- Sélection de la plate-forme et mises à jour de la chimie : Certaines plates-formes de séquençage et chimies sont plus sujettes au changement d’index que d’autres. Par exemple, les cellules d’écoulement à motifs et les technologies d’amplification d’exclusion ont été associées à des taux plus élevés de saut d’index. Rester informé des dernières améliorations des plates-formes et des lancements chimiques par les fabricants peut aider les laboratoires à sélectionner des systèmes avec des taux réduits de changement d’index.
Solutions bioinformatiques
- Algorithmes de démultiplexion rigoureux : Des outils de démultiplexion avancés peuvent être configurés pour exiger des correspondances parfaites avec les deux séquences d’index, rejetant les lectures avec des combinaisons d’index ambiguës ou inattendues. Cela réduit le risque que des lectures mal attribuées entrent dans les analyses ultérieures.
- Filtrage statistique et détection de contamination : Les pipelines bioinformatiques peuvent incorporer des modèles statistiques pour identifier et filtrer les combinaisons d’index à faible fréquence qui sont susceptibles d’être le résultat du changement d’index. Certains pipelines identifient également ou suppriment les lectures qui apparaissent dans des paires d’index inattendues, améliorant davantage la qualité des données.
- Évaluation de la contamination entre échantillons : L’évaluation régulière de la contamination croisée entre échantillons à l’aide de contrôles internes ou de spike-ins synthétiques peut aider à quantifier et à corriger les artefacts de changement d’index. Cela est particulièrement important dans des applications sensibles telles que le séquençage unicellulaire ou la détection de variantes rares.
En résumé, une combinaison de meilleures pratiques de laboratoire et d’approches bioinformatiques sophistiquées est nécessaire pour atténuer le changement d’index dans le séquençage ADN. La collaboration continue entre les fournisseurs de technologie de séquençage, tels qu’Illumina et Thermo Fisher Scientific, et la communauté scientifique continue de favoriser des améliorations tant dans la conception expérimentale que dans l’analyse des données, garantissant ainsi la fiabilité des données de séquençage multiplexé en 2025 et au-delà.
Normes et directives de l’industrie (ex. : Illumina, NIH)
Le changement d’index, également connu sous le nom de saut d’index, est un artefact technique bien reconnu dans le séquençage ADN à haut débit, en particulier dans les flux de travail de séquençage multiplexé. Ce phénomène se produit lorsque les indices d’échantillon (codes-barres) sont incorrectement attribués aux lectures de séquençage, conduisant à une mauvaise attribution des données entre les échantillons. À mesure que l’adoption du séquençage de nouvelle génération (NGS) s’est étendue à travers les applications de recherche, cliniques et industrielles, le besoin de normes et directives de l’industrie robustes pour atténuer et surveiller le changement d’index est devenu de plus en plus critique.
Les principaux fournisseurs de plates-formes de séquençage, tels qu’Illumina, ont joué un rôle central dans l’établissement des meilleures pratiques pour minimiser le changement d’index. Illumina, un leader mondial dans la technologie NGS, a publié des notes et des protocoles techniques abordant les causes du changement d’index, qui est particulièrement répandu sur les plates-formes de cellules d’écoulement à motifs et lors de l’utilisation de bibliothèques à index unique. Leurs recommandations incluent l’utilisation de stratégies d’indexation double uniques (UDI), qui emploient deux codes-barres indépendants par échantillon, réduisant considérablement le risque de mauvaise attribution. Illumina fournit également des ensembles d’index validés et des outils logiciels pour démultiplexer, conçus pour détecter et corriger d’éventuels événements de changement d’index.
En plus des lignes directrices des fabricants, des organisations scientifiques et réglementaires plus larges ont contribué à l’élaboration de normes. Les National Institutes of Health (NIH), en tant qu’agence de recherche biomédicale de premier plan aux États-Unis, a émis des recommandations pour les chercheurs utilisant le NGS dans des projets financés par le gouvernement fédéral. Le NIH encourage l’adoption de l’indexation double et de mesures rigoureuses de contrôle de la qualité, en particulier dans les études où la contamination croisée des échantillons pourrait compromettre l’intégrité des données ou la sécurité des patients. Ces recommandations sont souvent intégrées dans les exigences de subvention et les politiques de partage de données.
À l’international, des organisations telles que l’Organisation internationale de normalisation (ISO) ont développé des normes pour les pratiques de laboratoire en génomique, y compris l’ISO 20387 pour la biobanque et l’ISO 15189 pour les laboratoires médicaux. Bien qu’elles ne soient pas toujours spécifiques au changement d’index, ces normes mettent l’accent sur la traçabilité, la validation des méthodes et la documentation—des principes qui sous-tendent une détection et une atténuation efficaces de la mauvaise attribution d’index.
De plus, les sociétés professionnelles et les consortiums, y compris la Global Alliance for Genomics and Health (GA4GH), ont publié des cadres de meilleures pratiques pour la qualité des données NGS et le suivi des échantillons. Ces cadres font souvent référence aux protocoles des fabricants et aux recommandations réglementaires, favorisant l’harmonisation entre les laboratoires et les juridictions.
En résumé, les normes et lignes directrices industrielles pour traiter le changement d’index dans le séquençage ADN sont façonnées par une combinaison de protocoles des fabricants, de recommandations des agences de recherche nationales et de normes de laboratoire internationales. Le respect de ces lignes directrices est essentiel pour garantir l’exactitude des données, la reproductibilité et la fiabilité des analyses ultérieures tant dans les contextes de recherche que cliniques.
Marché et intérêt public : Tendances actuelles et prévisions sur 5 ans
Le changement d’index, également connu sous le nom de saut d’index, est un phénomène dans le séquençage ADN où les indices d’échantillon (codes-barres) sont incorrectement attribués aux lectures de séquençage, conduisant à une contamination croisée entre des échantillons multiplexés. Ce problème est particulièrement pertinent pour les plates-formes de séquençage à haut débit, telles que celles développées par Illumina, un leader mondial dans la technologie génomique. À mesure que l’adoption du séquençage de nouvelle génération (NGS) s’élargit à travers les diagnostics cliniques, la recherche et la biotechnologie, le marché et l’intérêt public pour traiter le changement d’index ont considérablement augmenté.
En 2025, le marché du séquençage ADN continue d’enregistrer une croissance robuste, alimentée par une demande croissante pour la médecine de précision, la génomique de population et la surveillance des maladies infectieuses. Le marché mondial de NGS devrait connaître un taux de croissance annuel composé (CAGR) dépassant 15 %, avec l’Amérique du Nord, l’Europe et l’Asie-Pacifique comme principales régions d’activité. Dans ce contexte, l’intégrité des données de séquençage est primordiale, et le changement d’index est devenu une préoccupation qualité critique. Les principaux fournisseurs de plates-formes de séquençage, notamment Illumina et Thermo Fisher Scientific, ont réagi en développant des kits de préparation de bibliothèque améliorés, des stratégies d’indexation double et des solutions logicielles pour atténuer le risque de mauvaise attribution d’index.
L’intérêt public pour la fiabilité des données de séquençage est également en hausse, surtout à mesure que les informations génomiques deviennent intégrales aux décisions de santé et aux politiques de santé publique. Les organismes réglementaires tels que la Food and Drug Administration (FDA) des États-Unis et des organisations internationales comme l’Organisation mondiale de la santé (OMS) portent une attention croissante aux normes et à la reproductibilité des tests génomiques, y compris l’impact des artefacts techniques comme le changement d’index. Cela a conduit à la publication de lignes directrices de bonnes pratiques et à l’incorporation de mesures de contrôle qualité dans les flux de travail de séquençage clinique.
En regardant vers les cinq prochaines années, la tendance est à une plus grande automatisation, un débit plus élevé et des multiplexages plus complexes dans les flux de travail de séquençage. Cela augmentera probablement le potentiel de changement d’index à moins d’être contrecarré par des innovations continues. Le marché devrait également voir de nouveaux investissements dans des chimies d’indexation robustes, des algorithmes de correction d’erreurs et des services de validation tiers. De plus, à mesure que le séquençage est adopté dans des contextes décentralisés et dans des points de soins, des solutions faciles à utiliser pour minimiser le changement d’index seront très demandées.
En résumé, le marché et l’intérêt public pour le changement d’index dans le séquençage ADN devraient s’intensifier d’ici 2030, alimentés par le rôle croissant de la génomique en médecine et en recherche. Les parties prenantes—y compris les développeurs de technologie, les agences de réglementation et les utilisateurs finaux—devraient prioriser des solutions garantissant la fidélité des données, soutenant ainsi la croissance continue et la confiance dans les applications basées sur le séquençage.
Perspectives d’avenir : Innovations, défis et voie à suivre
Le changement d’index, également connu sous le nom de saut d’index, reste une préoccupation majeure dans le séquençage ADN à haut débit, en particulier dans les expériences multiplexées où les échantillons sont regroupés et distingués par des séquences d’index uniques. À mesure que les technologies de séquençage avancent et que les applications s’étendent—du diagnostic clinique à la génomique de population à grande échelle—le besoin de traiter le changement d’index devient de plus en plus urgent. En regardant vers 2025, les perspectives futures pour gérer et atténuer le changement d’index sont façonnées à la fois par l’innovation technologique et des défis persistants.
L’un des domaines d’innovation les plus prometteurs est le développement de chimies et de plates-formes de préparation de bibliothèques améliorées. Les principaux fournisseurs de technologie de séquençage, tels qu’Illumina et Thermo Fisher Scientific, affinent activement leurs réactifs et protocoles pour minimiser le risque de mauvaise attribution d’index. Par exemple, l’adoption de stratégies d’indexation double uniques (UDI)—où deux séquences d’index indépendantes sont utilisées par échantillon—a déjà montré une réduction substantielle des événements de changement d’index. D’autres améliorations dans la synthèse et la purification des oligonucleotides sont attendues pour réduire le bruit de fond qui contribue à la mauvaise attribution.
Sur le plan computationnel, les outils bioinformatiques évoluent pour détecter et corriger plus efficacement le changement d’index. Des algorithmes qui modélisent la distribution attendue des combinaisons d’index et signalent les patterns anormaux sont intégrés dans les pipelines d’analyse standard des données de séquençage. Ces avancées sont soutenues par des efforts collaboratifs d’organisations telles que les National Institutes of Health (NIH), qui financent la recherche sur les solutions expérimentales et computationnelles pour les artefacts de séquençage.
Malgré ces avancées, plusieurs défis persistent. À mesure que le débit de séquençage augmente et que le multiplexage des échantillons devient plus courant, même de faibles taux de changement d’index peuvent avoir un impact significatif sur la qualité des données, en particulier dans les applications nécessitant une haute sensibilité, telles que la détection de variantes rares ou le séquençage unicellulaire. De plus, la diversité des plates-formes de séquençage et des chimies complique le développement de solutions universelles. La normalisation des meilleures pratiques à travers l’industrie, sous l’égide d’organismes tels que le National Human Genome Research Institute (NHGRI), sera cruciale pour garantir l’intégrité des données.
En regardant vers l’avenir, la voie pour minimiser le changement d’index impliquera probablement une combinaison de protocoles de laboratoire améliorés, de méthodes de correction computationnelles robustes et de normes à l’échelle de l’industrie. La collaboration continue entre les développeurs de technologie, les institutions de recherche et les agences réglementaires sera essentielle pour s’assurer que les bénéfices du séquençage à haut débit ne soient pas minés par des artefacts techniques. Alors que le domaine se dirige vers des projets de séquençage de plus en plus grands et complexes, traiter le changement d’index restera une priorité pour la communauté génomique.
Sources & Références
- National Institutes of Health
- Illumina
- Thermo Fisher Scientific
- Thermo Fisher Scientific
- National Institutes of Health
- Organisation internationale de normalisation
- Organisation mondiale de la santé
https://youtube.com/watch?v=WKAUtJQ69n8