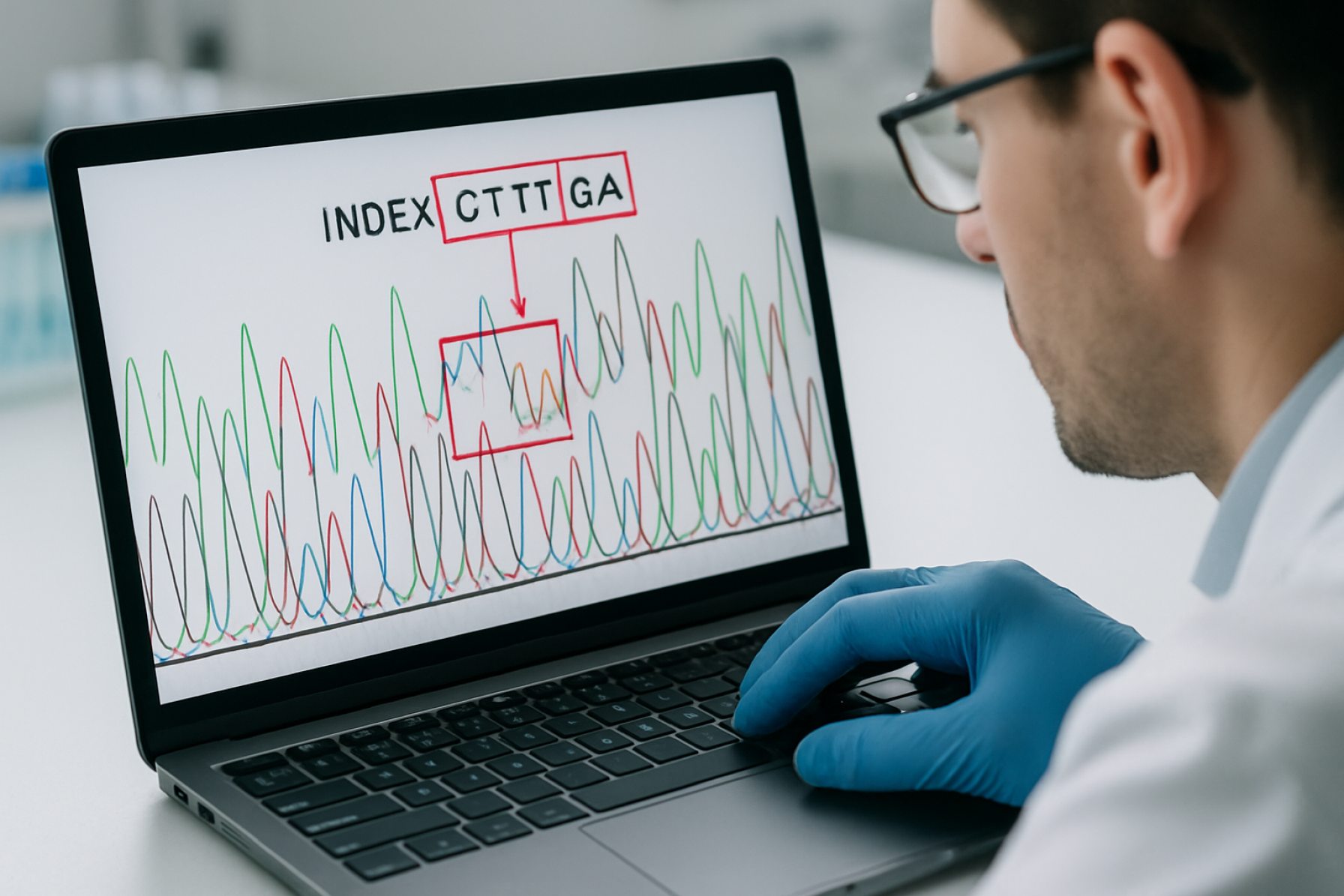
Index Switching bei der DNA-Sequenzierung: Die stille Bedrohung für die Datenintegrität und wie modernste Technologien zurückschlagen. Entdecken Sie die Auswirkungen, die Erkennung und die Zukunft dieses kritischen Phänomens. (2025)
- Einführung: Was ist Index Switching bei der DNA-Sequenzierung?
- Historischer Kontext und Entdeckung des Index Switching
- Mechanismen hinter dem Index Switching: Wie und warum es auftritt
- Technologische Faktoren: Sequenzierungsplattformen und Protokolle
- Folgen für die Genauigkeit genomischer Daten und Forschung
- Erkennungsmethoden: Identifizierung und Quantifizierung des Index Switching
- Minderungsstrategien: Labor- und bioinformatische Lösungen
- Branchenspezifische Standards und Richtlinien (z. B. Illumina, NIH)
- Markt- und öffentliches Interesse: Aktuelle Trends und 5-Jahres-Prognose
- Zukünftige Aussichten: Innovationen, Herausforderungen und der Weg nach vorne
- Quellen & Referenzen
Einführung: Was ist Index Switching bei der DNA-Sequenzierung?
Index Switching, auch bekannt als „Index Hopping“, ist ein Phänomen, das bei Hochdurchsatz-DNA-Sequenzierungsplattformen beobachtet wird, insbesondere bei solchen, die multiplexierte Sequenzierungsstrategien verwenden. Bei diesen Ansätzen werden einzigartige kurze DNA-Sequenzen – sogenannte Indizes oder Barcode – an individuelle DNA-Proben angeheftet. Dies ermöglicht es, mehrere Proben zu einem Pool zusammenzufassen und in einem einzigen Durchgang zu sequenzieren, mit der Erwartung, dass jeder Read später genau seiner ursprünglichen Probe anhand seines Index zugeordnet werden kann. Index Switching tritt jedoch auf, wenn die mit einem DNA-Fragment verbundene Indexsequenz fälschlicherweise zugeordnet wird, wodurch Reads fälschlicherweise der falschen Probe zugeschrieben werden.
Diese Fehlzuordnung kann in verschiedenen Phasen des Sequenzierungsablaufs auftreten, einschließlich der Bibliotheksvorbereitung, Clustererzeugung und der Sequenzierung selbst. Das Problem ist besonders ausgeprägt bei Plattformen, die gemusterte Flusszellen und Ausschlussamplifikation verwenden, wie z.B. die von Illumina, Inc., einem führenden Anbieter von Next-Generation Sequencing (NGS)-Technologie. In diesen Systemen können frei schwebende Adapter oder unvollständige Ligationsevents zu einem Transfer von Indexsequenzen zwischen DNA-Fragmenten führen, wodurch ein Teil der Reads den falschen Index trägt.
Die Folgen des Index Switching sind für viele Anwendungen der DNA-Sequenzierung erheblich. In Studien, in denen seltene Varianten oder Signale mit niedriger Häufigkeit wichtig sind – wie z.B. in der Einzelzellgenomik, Metagenomik oder klinischen Diagnostik – kann selbst eine geringe Rate an Indexfehlzuordnungen zu falsch positiven Ergebnissen, Kontamination oder fehlerhaften Schlussfolgerungen führen. Mit zunehmendem Sequenzdurchsatz und Multiplexing-Niveaus wird der potenzielle Einfluss des Index Switching auf die Datenqualität und -interpretation deutlicher.
Das Bewusstsein für Index Switching hat die Genomik-Gemeinschaft dazu veranlasst, sowohl experimentelle als auch rechnergestützte Strategien zur Minderung seiner Auswirkungen zu entwickeln. Dazu gehören der Einsatz von einzigartigen dualen Indizes (UDIs), verbesserte Protokolle zur Bibliotheksvorbereitung und bioinformatische Filtermethoden. Organisationen wie die National Institutes of Health (NIH) und National Human Genome Research Institute (NHGRI) haben die Bedeutung einer genauen Probenidentifikation in Sequenzierungsstudien hervorgehoben und die Notwendigkeit robuster Lösungen für das Index Switching unterstrichen.
Zusammenfassend lässt sich sagen, dass Index Switching eine kritische technische Herausforderung in der modernen DNA-Sequenzierung darstellt, mit Auswirkungen auf die Datenintegrität, Reproduzierbarkeit und die Zuverlässigkeit wissenschaftlicher und klinischer Erkenntnisse. Das Verständnis seiner Mechanismen und die Entwicklung effektiver Gegenmaßnahmen bleiben aktive Forschungs- und Innovationsbereiche in der Genomik.
Historischer Kontext und Entdeckung des Index Switching
Das Phänomen des Index Switching, auch bekannt als „Index Hopping“, trat in der Mitte der 2010er Jahre als bedeutendes Anliegen im Bereich der Hochdurchsatz-DNA-Sequenzierung hervor. Index Switching bezieht sich auf die Fehlzuordnung von Probenindizes (Barcodes) während frappentierter Sequenzierungsdurchläufe, wodurch Reads fälschlicherweise der falschen Probe zugeordnet werden. Dieses Artefakt kann die Genauigkeit nachgelagerter Analysen beeinträchtigen, insbesondere in Anwendungen, die hohe Sensitivität erfordern, wie z.B. Einzelzellgenomik und Metagenomik.
Der historische Kontext des Index Switching steht in engem Zusammenhang mit der schnellen Entwicklung von Next-Generation Sequencing (NGS)-Technologien. Frühe NGS-Plattformen, wie die von Illumina und Thermo Fisher Scientific, ermöglichten die gleichzeitige Sequenzierung mehrerer Proben, indem sie jedem Bibliothekselement einzigartige Indexsequenzen zuordneten. Dieser Multiplexansatz erhöhte den Durchsatz dramatisch und senkte die Kosten, führte jedoch auch zu neuen Fehlerquellen. Zunächst lag der Fokus darauf, Kreuzkontaminationen während der Bibliotheksvorbereitung und Sequenzierung zu minimieren. Als jedoch die Sequenzierungstiefe und Sensitivität verbessert wurden, begannen Forscher, unerwartete Muster von Read-Fehlzuordnungen zu beobachten, die sich nicht durch traditionelle Kontamination erklären ließen.
Die Entdeckung des Index Switching als distinct technisches Artefakt wurde erstmals systematisch im Jahr 2017 beschrieben, als Studien, die die gemusterten Flusszellenplattformen von Illumina (wie HiSeq 4000 und NovaSeq) verwendeten, erhöhte Raten von Indexfehlzuordnungen berichteten. Forscher stellten fest, dass die Verwendung von Ausschlussamplifikation (ExAmp)-Chemie, die die Brückenamplifikation in diesen neueren Plattformen ersetzt hatte, mit einer erhöhten Anzahl von Index Switching-Ereignissen verbunden war. Dies wurde auf die Anwesenheit frei schwebender Adapter und die physische Nähe von Clustern auf gemusterten Flusszellen zurückgeführt, die den Transfer von Indexsequenzen zwischen Bibliotheken während der Clustererzeugung erleichterten. Das Problem trat insbesondere in Einzelzell-RNA-seq-Experimenten auf, wo selbst geringe Mengen an Index Switching erhebliche Datenartefakte erzeugen konnten.
Als Reaktion auf diese Erkenntnisse erkannte die Sequenzierungstechnologieanbieter wie Illumina das Problem an und begann, bewährte Praktiken zur Minderung von Index Switching zu empfehlen, einschließlich der Verwendung einzigartiger dualer Indizes und verbesserter Protokolle zur Bibliotheksreinigung. Die breitere Genomik-Community, einschließlich Organisationen wie dem National Human Genome Research Institute (NHGRI), hat seitdem die Bedeutung des Verständnisses und der Kontrolle von Index Switching in der experimentellen Gestaltung und Dateninterpretation hervorgehoben. Bis 2025 läuft die laufende Forschung weiter, um Sequenzierungschemief und Informatikansätze zu verfeinern, um die Auswirkungen von Index Switching auf genomische Studien weiter zu reduzieren.
Mechanismen hinter dem Index Switching: Wie und warum es auftritt
Index Switching, auch bekannt als Index Hopping, ist ein Phänomen in der Hochdurchsatz-DNA-Sequenzierung, bei dem die spezifischen Indexsequenzen (Barcodes), die während der Bibliotheksvorbereitung zugewiesen werden, fälschlicherweise mit DNA-Fragmenten von anderen Proben assoziiert sind. Diese Fehlzuordnung kann zu einer Kreuzkontamination von Sequenzierungsreads führen, was potenziell nachgelagerte Analysen verwirren kann, insbesondere in multiplexen Experimenten, in denen viele Proben zusammengefasst werden.
Der primäre Mechanismus hinter dem Index Switching steht im Zusammenhang mit der Chemie und dem Arbeitsablauf von Sequenzierungsplattformen, insbesondere solchen, die gemusterte Flusszellen und Ausschlussamplifikation verwenden, wie etwa bestimmte Modelle von Illumina. Während der Bibliotheksvorbereitung werden einzigartige Indexsequenzen in DNA-Fragmenten ligiert oder integriert, um die Probenidentifikation nach der Sequenzierung zu ermöglichen. In einigen Fällen bleiben jedoch frei schwebende Adapter oder unvollständige Ligationprodukte in der gepoolten Bibliothek vorhanden. Während der Clustererzeugung auf der Flusszelle können diese freien Adapter an DNA-Fragmenten von verschiedenen Proben annealieren, was zur Incorporierung eines falschen Index während der Amplifikation führt. Dieser Prozess wird in Arbeitsabläufen verstärkt, die Ausschlussamplifikation verwenden, bei der DNA-Fragmenten immobilisiert und in enger Nähe amplifiziert werden, wodurch die Wahrscheinlichkeit einer Indexfehlzuordnung steigt.
Ein weiterer beitragender Faktor ist die Verwendung von kombinatorischem Dual-Indexing, bei dem zwei Indizes (i5 und i7) kombiniert werden, um die Multiplexkapazität zu erhöhen. Wenn Index Switching auftritt, kann ein Read eine Kombination von Indizes zugewiesen werden, die in der ursprünglichen Bibliothek nie vorhanden war, was es schwierig macht, den wahren Ursprung des Fragments nachzuvollziehen. Dies ist besonders problematisch in Anwendungen, die hohe Sensitivität erfordern, wie z.B. bei der Einzelzell-RNA-Sequenzierung, bei der selbst niedrige Niveaus von Index Switching erhebliche Artefakte einführen können.
Die Rate des Index Switching kann von verschiedenen Faktoren beeinflusst werden, darunter die Qualität der Bibliotheksvorbereitung, die Anwesenheit von überschüssigen Adaptern, die verwendete Sequenzierungsplattform und die spezifische Chemie der Flusszelle. Beispielsweise werden gemusterte Flusszellen, die dazu entworfen wurden, die Clusterdichte und den Durchsatz zu erhöhen, mit höheren Raten von Index Switching in Verbindung gebracht als nicht gemusterte Flusszellen. Außerdem kann die Verwendung von einzigartigen dualen Indizes (bei denen jeder Probe ein einzigartiges Paar von Indizes zugewiesen wird) helfen, die Auswirkungen von Index Switching zu mildern, indem es einfacher wird, falsch zugeordnete Reads zu identifizieren und herauszufiltern.
Das Verständnis der Mechanismen hinter dem Index Switching ist für Forscher und Sequenzierungsanbieter wie Illumina und Thermo Fisher Scientific von entscheidender Bedeutung, da dies die Entwicklung verbesserter Protokolle zur Bibliotheksvorbereitung und Sequenzierungschemien informiert. Laufende Forschung und technologische Fortschritte zielen darauf ab, das Index Switching zu minimieren und somit die Genauigkeit und Zuverlässigkeit von multiplexen DNA-Sequenzierungsexperimenten zu verbessern.
Technologische Faktoren: Sequenzierungsplattformen und Protokolle
Index Switching, auch bekannt als Index Hopping, ist ein Phänomen bei der DNA-Sequenzierung, bei dem Probenindizes (Barcodes) fälschlicherweise Sequenzierungsreads zugeordnet werden, was zu einer Fehlzuordnung von Sequenzen zwischen multiplexen Proben führt. Dieses Problem ist besonders relevant bei Hochdurchsatz-Sequenzierungsplattformen, die kombinatorische Barcode-Strategien verwenden, wie sie von Illumina, einem globalen Marktführer in der Next-Generation Sequencing (NGS)-Technologie, entwickelt wurden. Die technologischen Faktoren, die zum Index Switching beitragen, hängen eng mit dem Design der Sequenzierungsplattformen und den Protokollen zusammen, die während der Bibliotheksvorbereitung und Sequenzierungsdurchläufe verwendet werden.
Die Architektur der Sequenzierungsplattformen spielt eine kritische Rolle bei der Häufigkeit des Index Switching. Beispielsweise sind gemusterte Flusszellen, die in fortschrittlichen Illumina-Sequenzierern wie der NovaSeq-Serie verwendet werden, mit höheren Raten von Index Hopping verglichen mit früheren, nicht gemusterten Flusszellen-Designs assoziiert. Dies ist teilweise auf die physische Nähe von DNA-Clustern und die Verwendung von Ausschlussamplifikation zurückzuführen, die den Transfer von frei schwebenden Adaptern oder Indizes zwischen Clustern während des Sequenzierungsprozesses erleichtern kann. Die Chemie der Sequenzierungsreaktion, einschließlich der Verwendung bestimmter Polymerasen und der Anwesenheit überschüssiger Adapter, kann diesen Effekt weiter verstärken.
Die Protokolle zur Bibliotheksvorbereitung sind ein weiterer bedeutender technologischer Faktor. Strategien mit dualem Indexing, bei denen beide Enden des DNA-Fragments mit einzigartigen Indizes versehen werden, haben gezeigt, dass sie die Auswirkungen von Index Switching im Vergleich zu Methoden mit einfachem Indexieren reduzieren. Selbst bei dualem Indexing kann jedoch eine unvollständige Entfernung von freien Adaptern oder unangemessenen Reinigungsschritten dazu führen, dass Restindizes in der Reaktionsmischung verbleiben, was das Risiko einer Fehlzuordnung erhöht. Die Auswahl der Reagenzien, die Effizienz enzymatischer Reaktionen und die Strenge der Reinigungsschritte beeinflussen alle die Wahrscheinlichkeit von Index Switching-Ereignissen.
Der Sequenzierungsdurchsatz und die Multiplexniveaus beeinflussen ebenfalls die Raten des Index Switching. Wenn die Anzahl der in einem einzigen Sequenzierungsdurchgang gepoolten Proben zunimmt, steigt die Wahrscheinlichkeit einer Fehlzuordnung des Index, insbesondere wenn die Indizes nicht ausreichend einzigartig sind oder es während der Probenhandhabung zu Kontamination kommt. Dies ist ein besonderes Anliegen bei großangelegten genomischen Projekten und klinischen Anwendungen, bei denen eine genaue Probenidentifikation von entscheidender Bedeutung ist.
Um diesen Herausforderungen zu begegnen, haben Plattformhersteller wie Illumina und Forschungsverbünde bewährte Praktiken entwickelt, einschließlich der Verwendung einzigartiger dualer Indizes, rigoroser Reinigungsprotokolle für Bibliotheken und rechnergestützter Methoden zur Erkennung und Korrektur von Index Switching-Artefakten. Laufende technologischen Innovationen in der Sequenzierungschemie, dem Design von Flusszellen und der Automatisierung dürften die Auswirkungen des Index Switching im Jahr 2025 und darüber hinaus weiter mildern.
Folgen für die Genauigkeit genomischer Daten und Forschung
Index Switching, auch bekannt als Index Hopping, ist ein Phänomen bei der Hochdurchsatz-DNA-Sequenzierung, bei dem Probenindizes (Barcodes) fälschlicherweise Sequenzierungsreads zugeordnet werden. Diese Fehlzuordnung kann erhebliche Folgen für die Genauigkeit genomischer Daten und die Integrität der nachgelagerten Forschung haben. Da Sequenzierungsplattformen, insbesondere solche, die gemusterte Flusszellen und bestimmte Chemien zur Bibliotheksvorbereitung verwenden, zunehmend verbreitet werden, hat das Risiko und die Auswirkungen des Index Switching die Aufmerksamkeit der Genomik-Community auf sich gezogen.
Eine der wichtigsten Folgen des Index Switching ist die Einführung von Kreuzprobenkontamination. Wenn Reads fälschlicherweise der falschen Probe zugeschrieben werden, kann dies zu falsch positiven Ergebnissen führen – das Entdecken genetischer Varianten oder Sequenzen, die in einer bestimmten Probe nicht wahrhaftig vorhanden sind. Dies ist besonders problematisch in Studien, die sich mit seltenen Varianten, der Erkennung seltener Krankheitserreger oder mit Einzelzellsequenzierung befassen, bei denen selbst eine geringe Anzahl von fehlzugeordneten Reads die Ergebnisse verzerren und zu fehlerhaften biologischen Schlussfolgerungen führen kann. Zum Beispiel kann Index Switching in der Krebsgenomik zu einer Fehlidentifizierung somatischer Mutationen führen, was potenziell diagnostische oder therapeutische Entscheidungen beeinflussen kann.
Die Auswirkungen des Index Switching erstrecken sich auch auf großangelegte Bevölkerungsstudien und Metagenomik, bei denen eine genaue Demultiplexierung von Proben für eine zuverlässige Dateninterpretation unerlässlich ist. In metagenomischen Erhebungen kann Index Switching die Vielfalt mikrobieller Gemeinschaften künstlich erhöhen oder echte biologische Signale verdecken, was die Bemühungen zur Erforschung komplexer Ökosysteme erschwert. Ähnlich kann die Fehlallokation von Reads in der Populationsgenetik Analysen der genetischen Struktur, Abstammung und Assoziationsstudien verwirren und die Validität der Forschungsergebnisse untergraben.
Um diesen Herausforderungen zu begegnen, haben Sequenzierungstechnologieanbieter wie Illumina verbesserte Protokolle zur Bibliotheksvorbereitung und Strategien für duales Indexing entwickelt, um das Risiko des Index Switching zu minimieren. Dual Indexing, bei dem zwei einzigartige Barcodes pro Probe verwendet werden, reduziert signifikant die Wahrscheinlichkeit einer Fehlzuordnung, da beide Indizes gleichzeitig wechseln müssten, damit ein Fehler auftritt. Darüber hinaus werden immer häufiger bioinformatische Tools und Maßnahmen zur Qualitätskontrolle eingesetzt, um potenzielle Index-switched Reads zu erkennen und herauszufiltern, obwohl diese Ansätze das Problem möglicherweise nicht vollständig beseitigen können.
Die Folgen des Index Switching unterstreichen die Bedeutung eines rigorosen experimentellen Designs, der sorgfältigen Auswahl von Sequenzierungsplattformen und der Implementierung robuster Datenanalyse-Pipelines. Während das Gebiet der Genomik weiterhin Fortschritte macht, zielen laufende Bemühungen von Organisationen wie den National Institutes of Health und dem National Human Genome Research Institute darauf ab, bewährte Verfahren und Standards festzulegen, um die Genauigkeit und Reproduzierbarkeit genomischer Forschung angesichts technischer Herausforderungen wie Index Switching sicherzustellen.
Erkennungsmethoden: Identifizierung und Quantifizierung des Index Switching
Index Switching, auch bekannt als Index Hopping, ist ein Phänomen in der multiplexierten DNA-Sequenzierung, bei dem Probenindizes (Barcodes) fälschlicherweise Sequenzierungsreads zugeordnet werden, was zu einer Fehlzuordnung von Daten zwischen Proben führt. Die genaue Erkennung und Quantifizierung von Index Switching sind entscheidend, um die Datenintegrität zu gewährleisten, insbesondere in Anwendungen wie Metagenomik, Einzelzellsequenzierung und klinischen Diagnosen. Es wurden mehrere Erkennungsmethoden entwickelt, um Index Switching-Ereignisse zu identifizieren und zu quantifizieren, und nutzen sowohl experimentelles Design als auch computergestützte Analyse.
Ein grundlegender Ansatz zur Erkennung von Index Switching umfasst die Verwendung von negativen Kontrollen und synthetischen Spike-ins. Indem Proben mit einzigartigen, bekannten Sequenzen oder synthetischer DNA, die nicht mit biologischen Proben überlappen sollten, eingeschlossen werden, können Forscher auf unerwartete Indexkombinationen überwachen. Die Erkennung dieser unerwarteten Kombinationen in den Sequenzierungsdaten liefert eindeutige Beweise für das Index Switching. Diese Methode wird von Anbietern von Sequenzierungsplattformen wie Illumina, einem führenden Hersteller von Next-Generation Sequencing (NGS)-Instrumenten, empfohlen, die Leitlinien für das experimentelle Design zur Minimierung und Erkennung des Index Hoppings veröffentlicht haben.
Eine weitere gängige Strategie ist die Verwendung von dual-indexing-Schemas, bei denen jede Probe mit zwei einzigartigen Indizes (i5 und i7) versehen ist. Dieser Ansatz ermöglicht die Identifizierung von Index Switching, indem Indexpaare erkannt werden, die während der Bibliotheksvorbereitung nicht verwendet wurden. Computertools können dann die Häufigkeit dieser unerwarteten Indexpaare quantifizieren und eine Schätzung der Rate des Index Switching liefern. Das Dual-Indexing ist mittlerweile bei vielen Sequenzierungsabläufen Standardpraxis, wie von Organisationen wie Illumina und Thermo Fisher Scientific empfohlen, die beide bedeutende Anbieter von Sequenzierungsreagenzien und Plattformen sind.
Die bioinformatische Analyse spielt eine entscheidende Rolle bei der Erkennung und Quantifizierung von Index Switching. Algorithmen können Sequenzierungsdaten nach Reads mit Indexkombinationen durchsuchen, die mit keiner der erwarteten Probenzuweisungen übereinstimmen. Durch den Vergleich der beobachteten Verteilung von Indexpaaren mit der erwarteten Verteilung können Forscher die Rate und das Muster von Index Switching schätzen. Einige Pipelines integrieren auch statistische Modelle, um wahres Index Switching von Sequenzierungsfehlern oder Kreuzkontamination zu unterscheiden. Die National Institutes of Health (NIH), eine wichtige biomedizinische Forschungsagentur, hat die Entwicklung von Open-Source-Tools und bewährten Verfahren zur Analyse von multiplexierten Sequenzierungsdaten unterstützt und dabei die Bedeutung robuster rechnergestützter Erkennungsmethoden betont.
Zusammenfassend lassen sich die Erkennung und Quantifizierung von Index Switching in der DNA-Sequenzierung auf eine Kombination aus experimentellen Kontrollen, Dual-Indexing-Strategien und fortschrittlichen bioinformatischen Analysen zurückführen. Die Einhaltung der von führenden Organisationen und Anbietern von Sequenzierungstechnologien empfohlenen bewährten Praktiken ist entscheidend, um die Auswirkungen des Index Switching zu minimieren und die Zuverlässigkeit der Sequenzierungsergebnisse sicherzustellen.
Minderungsstrategien: Labor- und bioinformatische Lösungen
Index Switching, auch bekannt als Index Hopping, ist ein gut dokumentiertes Artefakt bei der Hochdurchsatz-DNA-Sequenzierung, insbesondere in multiplexen Experimenten, bei denen mehrere Proben gepoolt und durch einzigartige Indexsequenzen unterschieden werden. Dieses Phänomen kann zu Fehlzuordnungen von Reads führen und die Datenintegrität und nachgelagerte Analysen gefährden. Angesichts der wachsenden Sequenzierungstechnologien und -anwendungen im Jahr 2025 sind robuste Minderungsstrategien – sowohl auf Labor- als auch auf bioinformatischer Ebene – entscheidend, um die Daten genau zu halten.
Laborlösungen
- Dual-Indexing: Eine der effektivsten Laborstrategien ist die Verwendung einzigartiger dualer Indizes (UDI), bei der jede Probe mit zwei unterschiedlichen Indexsequenzen versehen wird. Diese Methode reduziert signifikant die Wahrscheinlichkeit einer Fehlzuordnung, da beide Indizes gleichzeitig wechseln müssten, damit ein Read fälschlicherweise zugewiesen wird. Führende Anbieter von Sequenzierungsplattformen, wie Illumina, haben UDI-Kits und Protokolle integriert, um dieses Problem anzugehen.
- Optimierte Bibliotheksvorbereitung: Eine sorgfältige Optimierung der Bibliotheksvorbereitungsprotokolle kann die Kontamination durch freie Adapter minimieren, die eine bekannte Ursache für das Index Switching ist. Dazu gehören gründliche bead-basierte Reinigungen und enzymatische Entfernung von überschüssigen Adaptern. Organisationen wie Thermo Fisher Scientific bieten Leitlinien und Reagenzien an, um diese besten Praktiken zu unterstützen.
- Plattformauswahl und Chemie-Updates: Einige Sequenzierungsplattformen und Chemien sind anfälliger für Index Switching als andere. Beispielsweise wurden gemusterte Flusszellen und Technologien zur Ausschlussamplifikation mit höheren Raten von Index Hopping in Verbindung gebracht. Das Verfolgen der neuesten Plattformverbesserungen und Chemiefreigaben von Herstellern kann Laboren helfen, Systeme mit reduzierten Raten des Index Switching auszuwählen.
Bioinformatische Lösungen
- Strenge Demultiplexing-Algorithmen: Fortschrittliche Demultiplexing-Tools können so konfiguriert werden, dass sie perfekte Übereinstimmungen mit beiden Indexsequenzen erfordern, wobei Reads mit mehrdeutigen oder unerwarteten Indexkombinationen verworfen werden. Dies reduziert das Risiko, dass falsch zugeordnete Reads in nachgelagerte Analysen einfließen.
- Statistische Filterung und Kontaminationsdetecting: Bioinformatische Pipelines können statistische Modelle integrieren, um niedrigfrequente Indexkombinationen zu identifizieren und herauszufiltern, die wahrscheinlich das Ergebnis von Index Switching sind. Einige Pipelines kennzeichnen oder entfernen zudem Reads, die in unerwarteten Indexpaaren erscheinen, was die Datenqualität weiter verbessert.
- Bewertung der Kreuzprobenkontamination: Die regelmäßige Überprüfung der Kreuzprobenkontamination unter Verwendung interner Kontrollen oder synthetischer Spike-ins kann helfen, Index Switching-Artefakte zu quantifizieren und zu korrigieren. Dies ist besonders wichtig in sensiblen Anwendungen wie der Einzelzellsequenzierung oder der Erkennung seltener Varianten.
Zusammenfassend ist eine Kombination aus laborbasierten Best Practices und ausgeklüftelten bioinformatischen Ansätzen erforderlich, um das Index Switching in der DNA-Sequenzierung zu mindern. Die laufende Zusammenarbeit zwischen Anbietern von Sequenzierungstechnologien, wie Illumina und Thermo Fisher Scientific, und der wissenschaftlichen Gemeinschaft trägt weiterhin dazu bei, die Verbesserungen sowohl im experimentellen Design als auch in der Datenanalyse voranzutreiben, um die Zuverlässigkeit der multiplexen Sequierungsdaten im Jahr 2025 und darüber hinaus zu gewährleisten.
Branchenspezifische Standards und Richtlinien (z. B. Illumina, NIH)
Index Switching, auch bekannt als Index Hopping, ist ein gut anerkanntes technisches Artefakt in der Hochdurchsatz-DNA-Sequenzierung, insbesondere bei multiplexierten Sequenzierungsabläufen. Dieses Phänomen tritt auf, wenn Probenindizes (Barcodes) fälschlicherweise Sequenzierungsreads zugeordnet werden, was zu einer Fehlzuordnung von Daten zwischen Proben führt. Da die Anwendung von Next-Generation Sequencing (NGS) in der Forschung, Klinik und Industrie zugenommen hat, ist der Bedarf an robusten branchenspezifischen Standards und Richtlinien zur Minderung und Überwachung des Index Switching zunehmend kritisch geworden.
Wichtige Anbieter von Sequenzierungsplattformen, wie Illumina, haben eine zentrale Rolle bei der Festlegung von bewährten Praktiken zur Minimierung von Index Switching gespielt. Illumina, ein globaler Marktführer in der NGS-Technologie, hat technische Hinweise und Protokolle veröffentlicht, die die Ursachen für das Index Switching behandeln, das insbesondere in Plattformen mit gemusterten Flusszellen und bei der Verwendung von einfach-indexierten Bibliotheken weit verbreitet ist. Ihre Empfehlungen umfassen die Verwendung einzigartiger dualer Indexierungsstrategien (UDI), die zwei unabhängige Barcodes pro Probe verwenden, was das Risiko einer Fehlzuordnung erheblich reduziert. Illumina bietet auch validierte Indexsets und Softwaretools für die Demultiplexierung an, die dazu entwickelt wurden, um potenzielle Index Switching-Ereignisse zu erkennen und zu korrigieren.
Neben den Herstellerleitlinien haben auch breitere wissenschaftliche und regulatorische Organisationen zur Entwicklung von Standards beigetragen. Die National Institutes of Health (NIH), als führende biomedizinische Forschungsagentur in den Vereinigten Staaten, hat Leitlinien für Forscher veröffentlicht, die NGS in staatlich geförderten Projekten verwenden. Die NIH fördert die Übernahme von Dual-Indexing und rigorosen Qualitätskontrollmaßnahmen, insbesondere in Studien, bei denen die Kreuzkontamination von Proben die Datenintegrität oder die Patientensicherheit gefährden könnte. Diese Empfehlungen werden häufig in Anforderungen für Forschungsanträge und Datenfreigabepolitiken übernommen.
International haben Organisationen wie die International Organization for Standardization (ISO) Standards für Laborpraktiken in der Genomik entwickelt, einschließlich ISO 20387 für Biobankierung und ISO 15189 für medizinische Labors. Während nicht immer spezifisch für Index Switching, betonen diese Standards Rückverfolgbarkeit, Validierung von Methoden und Dokumentation – Prinzipien, die einer effektiven Erkennung und Minderung von Indexfehlzuordnungen zugrunde liegen.
Darüber hinaus haben Berufsverbände und Konsortien, darunter die Global Alliance for Genomics and Health (GA4GH), beste Praxisrahmen für die NGS-Datenqualität und Probenverfolgung veröffentlicht. Diese Rahmenwerke beziehen oft auf Herstellerprotokolle und regulatorische Richtlinien und fördern die Harmonisierung über Labore und Gerichtsbarkeiten hinweg.
Zusammenfassend werden die branchenspezifischen Standards und Richtlinien zur Behandlung des Index Switching in der DNA-Sequenzierung durch eine Kombination von Herstellerprotokollen, Empfehlungen nationaler Forschungsagenturen und internationalen Laborstandards geprägt. Die Einhaltung dieser Richtlinien ist entscheidend, um die Genauigkeit, Reproduzierbarkeit und Zuverlässigkeit nachgelagerter Analysen in sowohl Forschungs- als auch klinischen Umgebungen sicherzustellen.
Markt- und öffentliches Interesse: Aktuelle Trends und 5-Jahres-Prognose
Index Switching, auch bekannt als Index Hopping, ist ein Phänomen bei der DNA-Sequenzierung, bei dem Probenindizes (Barcodes) fälschlicherweise Sequenzierungsreads zugeordnet werden, was zu Kreuzkontamination zwischen multiplexen Proben führt. Dieses Problem ist besonders relevant bei Hochdurchsatz-Sequenzierungsplattformen, wie den von Illumina, einem globalen Marktführer in der Genomik-Technologie, entwickelten. Mit der Zunahme der Anwendung von Next-Generation Sequencing (NGS) in der klinischen Diagnostik, Forschung und Biotechnologie hat das Markt- und öffentliche Interesse an der Lösung des Index Switching erheblich zugenommen.
Im Jahr 2025 erfährt der Markt für DNA-Sequenzierung ein robustes Wachstum, angetrieben durch die zunehmende Nachfrage nach Präzisionsmedizin, Bevölkerungsgenomik und der Überwachung von Infektionskrankheiten. Der globale NGS-Markt wird voraussichtlich mit einer jährlichen Wachstumsrate (CAGR) von über 15 % expandieren, wobei Nordamerika, Europa und der asiatisch-pazifische Raum wichtige Regionen sind. In diesem Kontext ist die Integrität von Sequenzierungsdaten von größter Bedeutung, und Index Switching ist zu einem kritischen Qualitätsanliegen geworden. Wichtige Anbieter von Sequenzierungsplattformen, darunter Illumina und Thermo Fisher Scientific, haben darauf reagiert, indem sie verbesserte Kits zur Bibliotheksvorbereitung, Strategien für duales Indexing und Softwarelösungen entwickelt haben, um das Risiko von Indexfehlzuordnungen zu mindern.
Das öffentliche Interesse an der Zuverlässigkeit von Sequenzierungsdaten nimmt ebenfalls zu, insbesondere da genomische Informationen einen integralen Bestandteil von Gesundheitsentscheidungen und der öffentlichen Gesundheitspolitik bilden. Regulierungsbehörden wie die U.S. Food and Drug Administration und internationale Organisationen wie die Weltgesundheitsorganisation schenken den Standards und der Reproduzierbarkeit genomischer Assays zunehmend Beachtung, einschließlich der Auswirkungen technischer Artefakte wie Index Switching. Dies hat zur Veröffentlichung von Leitlinien für bewährte Praktiken und zur Integration von Qualitätskontrollmetriken in klinischen Sequenzierungsabläufen geführt.
Bei einem Ausblick auf die nächsten fünf Jahre wird der Trend in Richtung größerer Automatisierung, höherem Durchsatz und komplexeren Multiplexierungen in Sequenzierungsabläufen gehen. Dies wird wahrscheinlich das Potenzial für Index Switching erhöhen, es sei denn, es wird durch fortwährende Innovationen entgegengewirkt. Der Markt wird voraussichtlich weitere Investitionen in robuste Indexierungschemien, Fehlerkorrekturalgorithmen und Drittanbieter-Validierungsdienste sehen. Darüber hinaus werden in dezentralen und Point-of-Care-Einstellungen benutzerfreundliche Lösungen zur Minimierung von Index Switching stark nachgefragt werden.
Zusammenfassend werden das Markt- und öffentliche Interesse am Index Switching in der DNA-Sequenzierung bis 2030 zunehmen, angetrieben durch die wachsende Rolle der Genomik in der Medizin und Forschung. Interessengruppen – einschließlich Technologieentwickler, Regulierungsbehörden und Endnutzer – werden voraussichtlich Lösungen priorisieren, die die Datenintegrität gewährleisten und somit das kontinuierliche Wachstum und das Vertrauen in sequenzierungsbasierte Anwendungen unterstützen.
Zukünftige Aussichten: Innovationen, Herausforderungen und der Weg nach vorne
Index Switching, auch bekannt als Index Hopping, bleibt ein bedeutendes Anliegen in der Hochdurchsatz-DNA-Sequenzierung, insbesondere in multiplexen Experimenten, bei denen Proben gepoolt und durch einzigartige Indexsequenzen unterschieden werden. Mit dem Fortschritt der Sequenzierungstechnologien und der Expansion der Anwendungen – von klinischen Diagnosen bis zur großangelegten Bevölkerungsgenomik – wird die Notwendigkeit, das Index Switching zu adressieren, zunehmend dringlich. Wenn wir auf 2025 blicken, werden die zukünftigen Aussichten für das Management und die Minderung des Index Switching sowohl von technologischen Innovationen als auch von anhaltenden Herausforderungen geprägt.
Einer der vielversprechendsten Innovationsbereiche ist die Entwicklung verbesserter Chemien zur Bibliotheksvorbereitung und Sequenzierungsplattformen. Wichtige Anbieter von Sequenzierungstechnologie, wie Illumina und Thermo Fisher Scientific, verfeinern aktiv ihre Reagenzien und Protokolle, um das Risiko von Indexfehlzuordnungen zu minimieren. Beispielsweise hat die Einführung von einzigartigen dualen Indexierungsstrategien (UDI), bei denen zwei unabhängige Indexsequenzen pro Probe verwendet werden, bereits eine erhebliche Reduktion der Index Switching-Ereignisse gezeigt. Weitere Verbesserungen in der Oligonukleotidsynthese und -reinigung dürften das Hintergrundrauschen weiter vermindern, das zu Fehlzuordnungen führt.
Auf der rechnergestützten Seite entwickeln sich bioinformatische Tools weiter, um das Index Switching besser zu erkennen und zu korrigieren. Algorithmen, die die erwartete Verteilung von Indexkombinationen modellieren und anomale Muster kennzeichnen, werden in die Standardpipelines zur Datenanalyse bei Sequenzierungsdaten integriert. Diese Fortschritte werden durch gemeinsame Bemühungen von Organisationen wie den National Institutes of Health (NIH) unterstützt, die Forschung sowohl in experimentellen als auch in rechnergestützten Lösungen für Sequenzierungsartefakte finanzieren.
Trotz dieser Fortschritte bestehen weiterhin mehrere Herausforderungen. Mit steigender Sequenzierungsdurchsatz und zunehmendem Multiplexing, können selbst niedrige Raten von Index Switching erhebliche Auswirkungen auf die Datenqualität haben, insbesondere in Anwendungen, die eine hohe Sensitivität erfordern, wie z.B. die Erkennung seltener Varianten oder die Einzelzellsequenzierung. Darüber hinaus erschwert die Vielfalt von Sequenzierungsplattformen und -chemien die Entwicklung universeller Lösungen. Die Standardisierung von bewährten Praktiken in der Branche, geleitet von Institutionen wie dem National Human Genome Research Institute (NHGRI), wird entscheidend sein, um die Datenintegrität sicherzustellen.
In naher Zukunft wird der Weg zur Minimierung von Index Switching wahrscheinlich eine Kombination aus verbesserten Laborprotokollen, robusten rechnergestützten Korrekturmethoden und branchenweiten Standards umfassen. Die fortlaufende Zusammenarbeit zwischen Technologieentwicklern, Forschungseinrichtungen und Regulierungsbehörden wird entscheidend sein, um sicherzustellen, dass die Vorteile der Hochdurchsatz-Sequenzierung nicht durch technische Artefakte untergraben werden. Während das Gebiet auf immer größere und komplexere Sequierungsprojekte zuschreitet, wird die Auseinandersetzung mit dem Index Switching nach wie vor eine Priorität für die Genomik-Community bleiben.
Quellen & Referenzen
- National Institutes of Health
- Illumina
- Thermo Fisher Scientific
- Thermo Fisher Scientific
- National Institutes of Health
- International Organization for Standardization
- World Health Organization
https://youtube.com/watch?v=WKAUtJQ69n8